
비세포 유기체 (virusoids, viroids and viruses) 외에도 세포 유기체는 6개 계 (kingdoms) (bacteria, protozoa, chromista, plantae, fungi and animalia)으로 분류될 수 있으며, 이는 두 empires (prokaryota-원핵생물과 eukaryota-진핵생물)에 해당한다 (Cavalier-Smith, 2004). 비세포 개체, 단세포 및 다세포 생물의 유전자형 (genotype) (structural genomics)에 따라 표현형(phenotype)이 결정되며, 이는 결국 의인관(anthropomorphic) 및 생명공학적 관점에서 잠재적으로 유익할 수도 있고 위험할 수도 있다. 실제로, 표현형(phenotype)은 게놈의 발현 (expression of the genome) (functional genomics)과 환경 사이의 상호작용에 의해 생성된다.
반면, 유전자 발현 (gene expression)은 유전체 서열 (genome sequence), 후생유전적 요인(epigenetic factors), 환경적 요인에 의해 결정된다. 따라서 게놈 서열 결정과 추정되는 후성유전학적 변형(epigenetic modifications)을 결정하는 것이 가장 중요하다. 왜냐하면 유전자 발현과 따라서 표현형에 주요 관련성을 가질 수 있기 때문이다. 마찬가지로, 조직 특이적일 수도 있고 심지어 세포 특이적일 수도 있는 종의 전사체 서열 (transcriptome sequence)을 아는 것도 중요하다. 이런 지식은 생명공학에 중요한 응용 분야를 가지고 있다.
DNA의 효율적인 시퀀싱(서열 분석)은 시간이 많이 걸리는 단계와 방사성 동위원소를 포함한 독성 물질의 사용을 포함하여 처음에는 상당히 힘든 다양한 방법을 사용하여 많은 시도 끝에 달성되었다. 이 방법은 나중에 무독성 화학물질, 자동화, 소형화 및 높은 처리량 장비를 사용하여 개선되었다. 예전 방법론의 주요 한계 중 하나는 분석을 위해 대량의 동일한 분자(수천에서 수백만 개)가 필요하다는 것이었다.
이를 위해서는 먼저 생체 내에서 분자 복제 (molecular cloning) 증폭(amplifications)이 필요했고 나중에 중합효소 연쇄 반응 (PCR, Polymerase Chain Reaction)을 통해 시험관 내 증폭도 필요했다. 결국 단일 분자 서열 분석 접근법이 개발되었으며, 이는 마이크로칩 기술 및 대규모 병렬 생물정보학 분석과 함께 이 매력적인 기술의 새로운 최첨단을 나타낸다.
반면, 때로는 ‘차세대 Next-Generation’, 심지어 ‘차차세대 Next-Next-Generation’ 시퀀싱이라고도 불리는 다양한 DNA 서열 분석 기술의 이름을 지정하는 데 의미상 모호함과 혼란이 있다는 점에 유의해야 한다 (각각 NGS와 NNGS). 이는 매우 일반적이고 이름에 레퍼런스가 없으므로 몇 년 후에는 큰 의미가 없을 수 있다. 이를 방지하기 위해 여기서는 DNA 서열 분석 방법론을 세 가지 범주(즉, 각각 First-, Second- 그리고 Third-Generation Sequencing; FGS, SGS and TGS)로 나누는 관점을 따른다 (Clarke et al., 2009; Schadt et al., 2010; Schatz et al., 2011).

1세대 DNA 서열 분석 방법론에는 많은 양의 각 핵산 분자를 읽어야 하는 전략 (따라서 판독하려면 각 핵산 분자의 생체내 복제 (in vivo cloning) 또는 시험관내 증폭(in vitro amplification)이 필요)이 포함되어 있어 최근의 어푸로치들과 비교해볼 때 낮은 처리량을 생성한다. 일반적으로 각 실험 실행에서는 사용되는 구체적인 기술에 따라 겔 레인 (gel lane) 또는 모세관 (capillary) 당 최대 약 1,000개의 뉴클레오티드 염기에 대한 소수의 판독 값들만 생성한다. 즉, 일반적으로 매우 짧은 길이의 핵산 서열을 분석하는 데 사용된다. 그럼에도 불구하고, 이 어푸로치는 최적화되고 확장될 수 있으며, 아래에 표시된 대로 큰 게놈도 시퀀싱할 수 있는 반자동 기계를 사용하여 시간이 지남에 따라 개선될 수 있다.
연대순 관점에서 볼 때, 핵산에 대한 효율적인 서열분석은 많은 실험적 시도를 거쳐 달성되었다. 따라서 1973년에 Gilbert & Maxam은 'wandering-spot’ analysis methodology를 사용하여 the lac operator의 뉴클레오티드 서열 (단지 24개 뉴클레오티드)을 보고했다 (Gilbert and Maxam, 1973).
한편, Sanger & Coulson은 1975년에 획기적인 'plus & minus' 염기서열 분석 방법을 보고했는데 (Sanger and Coulson, 1975), 이 방법은 single-stranded DNA (ssDNA) 마련을 위해 생체 내 분자 복제가 필요했지만 당시 매우 인기가 있었다.
최초의 (스몰) 게놈은 1976년 Fiers et al.에 의해 서열 분석되었으며, 이는 MS2 박테리오파지(bacteriophage) (3,569개의 뉴클레오티드)에 해당하며 RiboNucleic Acid (RNA)이다 (Fiers et al., 1976).
두 번째 (스몰) 게놈은 그 다음에 Sanger et al.에 의해 서열 분석되었으며, 이는 Phi-X174 박테리오파지 DeoxyriboNucleic Acid (DNA)의 5,386개 뉴클레오티드에 상당하는 것이었다 (Sanger et al., 1977a).
1세대 핵산 서열 분석의 두 가지 주요 방법론은 아래에 설명되어 있다 (Maxam-Gilbert 및 Sanger).
1977년에 DNA 염기서열 분석을 위한 두 가지 선구적인 방법이 보고되었다. 하나는 Alan Maxam & Walter Gilbert에 의해, 다른 하나는 Frederick Sanger와 동료들에 의해 보고되었다. 그때까지는 5~10개 염기 길이의 짧은 핵산 가닥이라도 서열 분석이 어렵고 힘든 과정이었다. 핵산 합성 방법의 진보, 제한 효소의 발견, 핵산의 겔 전기영동 개발은 이 두 가지 획기적인 서열 분석 기술의 발명에 도움이 되었다. Maxam-Gilbert와 Sanger 시퀀싱 방법은 서로 다른 화학을 사용하지만 두 방법 모두 겔 전기영동과 결합된 방사성 표지를 통해 DNA 단편을 시각화하는 데 의존한다.


Maxam-Gilbert Sequencing 방법에서는 5' 말단이 방사성 표지된 DNA 단편이 서로 다른 뉴클레오티드(예: A와 G, G, C와 T 또는 C)에서 화학적으로 절단된다. 절단된 조각은 겔 전기영동으로 분리되고 자동 방사선 촬영으로 검출된다 (Figure 1).
Maxam-Gilbert Sequencing은 1976-1977년에 Allan Maxam & Walter Gilbert에 의해 개발된 DNA 시퀀싱 방법이다.
이 방법은 DNA의 핵염기-특유 부분적 화학 변화 (nucleobase-specific partial chemical modification of DNA)와 후속적인 변화된 뉴클레오티드들에 인접한 부위들에서의 DNA 백본의 분열(cleavage of the DNA backbone)에 기초한다.
Maxam-Gilbert 시퀀싱은 최초로 널리 채택된 DNA 서열분석 방법이었으며, Sanger dideoxy 방법과 함께 1세대 DNA 서열분석 방법을 대표한다. Maxam-Gilbert 시퀀싱은 차세대 시퀀싱 방법으로 대체되어 더 이상 널리 사용되지 않는다.
Maxam & Gilbert는 Frederick Sanger & Alan Coulson이 plus-minus 시퀀싱에 대한 연구를 발표한지 2년 후에 화학적 시퀀싱 방법을 발표했지만, 정제된 DNA를 직접 사용할 수 있기 때문에 Maxam-Gilbert 시퀀싱은 급속히 대중화되었다. 초기 Sanger 방법에서는 단일 가닥 (single-stranded) DNA 생산을 위해 각 리드 스타트가 클로닝 되어야 했다. 즉, 정제된 DNA를 직접 시퀀싱할 수 있고, in vivo cloning와 ssDNA 준비단계가 없어도 되므로 획기적인 방법으로 대두되었다.
그러나 사슬 종결 방법(chain-termination method)이 개선됨에 따라 Maxam-Gilbert 염기서열분석은 표준 분자생물학 키트에서 사용할 수 없는 기술적 복잡성, 위험한 화학물질의 광범위한 사용 및 규모 확장의 어려움으로 인해 선호되지 않게 되었다. 따라서 결국 더 깨끗하고 편리한 대안으로 대체되었다. 즉, Sanger 접근 방식이다.
Allan Maxam & Walter Gilbert의 1977 논문 “A new method for sequencing DNA”은 2017년 미국 화학학회 (the American Chemical Society) 화학사 부문에서 화학 혁신상(Chemical Breakthrough Award) 표창을 받았다. 이 논문은 하버드 대학교 분자 및 세포 생물학과에 제출되었다. Walter Gilbert는 Frederick Sanger 및 Paul Berg와 함께 1980년 노벨 화학상을 수상했다. Gilbert와 Sanger는 핵산의 뉴클레오티드 서열을 결정하는 방법을 고안하는 선구적인 연구로 인정을 받은 것이었다.
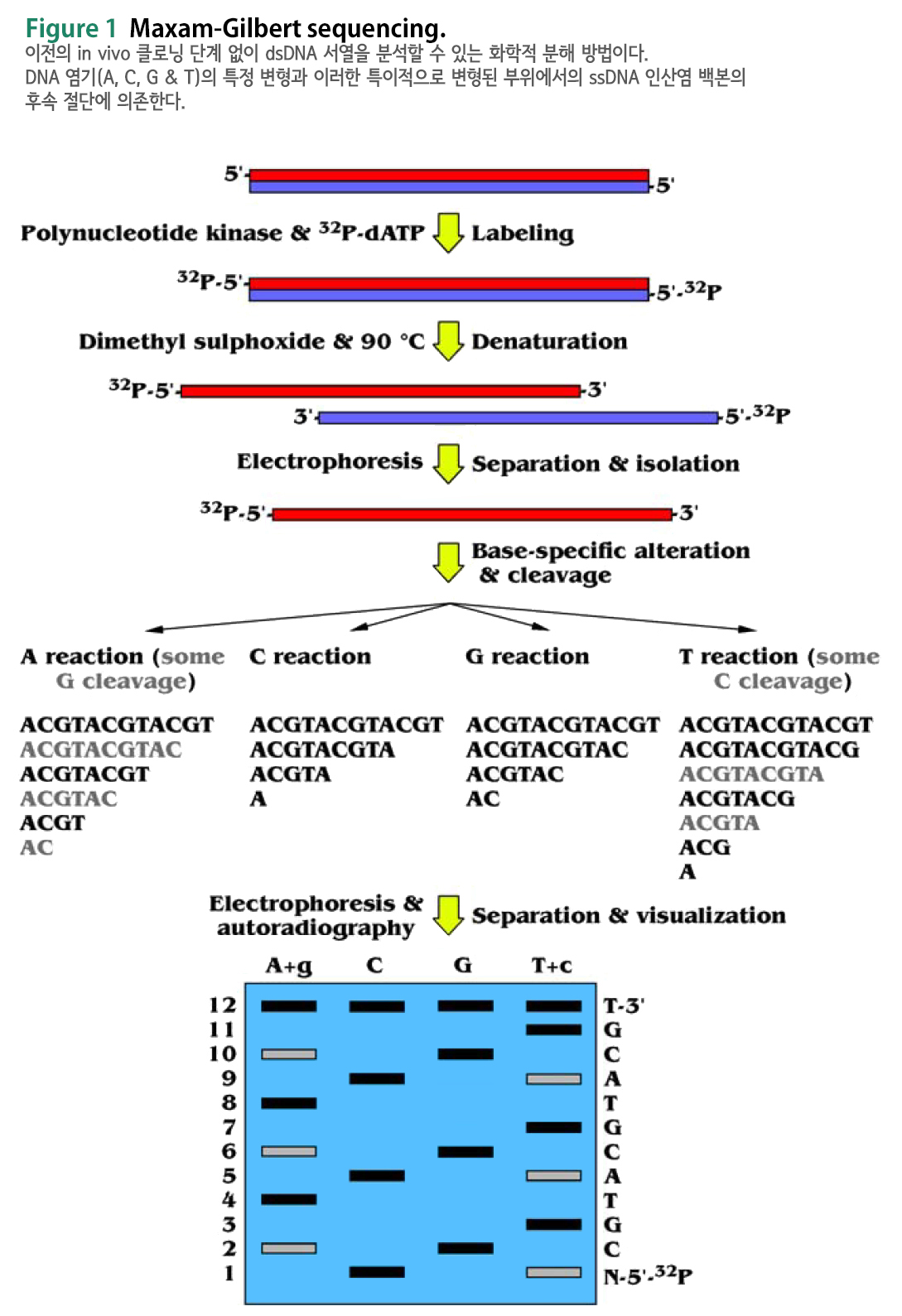
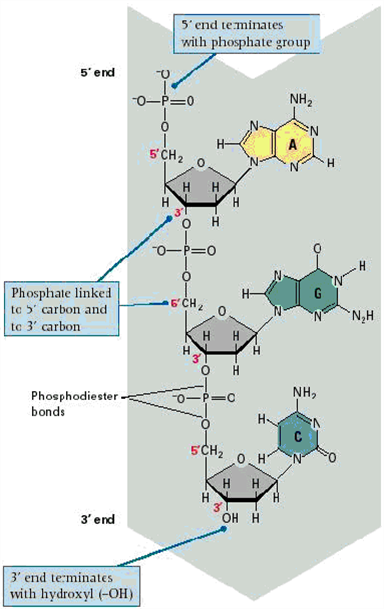
Maxam-Gilbert 시퀀싱에서는 시퀀스되어야 할 DNA fragment (단편)의 한쪽 5' 말단에 방사성 표지를 부착 (radioactive labeling)하고 (일반적으로 gamma-32P ATP를 사용하는 키나제 반응 (kinase reaction)에 의해), 그리고 DNA의 정제 (purification of the DNA)가 필요하다. 즉, double-stranded DNA (dsDNA)의 5′-P ends를 radioactive labeling (polynucleotide kinase를 사용하여 32P-dATP로 표지)한다.
그런 다음, 그 DNA는 DiMethyl SulfOxide (DMSO)로 90°C 에서 변성되고(denatured), 결과적인 ssDNA 분자들은 전기영동에 의해 분리된다 (segregated). Adenosine (A), Cytidine (C), Guanosine (G) 그리고 Thymidine (T) 잔기들을 변형하기 위해 질소 염기 특이적 반응들(Nitrogenous base-specific reactions)이 수행되어, 해당 위치의 5'-P 측에서 ssDNA의 화학적 절단이 가능해진다. A와 T 반응은 또한 각각 약간의 G와 C 절단(cleavage)을 생성하는데, 이는 고려되어야 한다 (나중에 더 약한 신호로 표시됨).
화학적 처리는 4가지 반응 각각에서 4개의 뉴클레오티드 염기들 중 1개 또는 2개 (G, A+G, C, C+T)의 작은 비율에서 파손 (breaks)을 생성한다. 예를 들어,
⊙ 퓨린(purines)(A+G)은 포름산(formic acid)을 사용하여 탈퓨린화되고 (depurinated),
⊙ 구아닌 (guanines) (그리고 어느 정도 아데닌 adenines)은
디메틸 황산(dimethyl sulfate)으로 메틸화되며 (methylated),
⊙ 피리미딘 (pyrimidines)(C+T)은 히드라진(hydrazine)을 사용하여 가수분해된다 (hydrolysed).
⊙ 히드라진 반응에 소금 (염화나트륨 sodium chloride)을 첨가하면 C-단독 반응에서 티민(thymine)의 반응이 억제된다.
변형된(modified) DNAs는 뜨거운 피페리딘(piperidine)에 의해 쪼개질 수 있다(be cleaved) ; 변형된 염기 위치에서의 (CH2)5NH. 변형시키는 화학물질의 농도는 DNA 분자당 평균 하나의 변형이 도입되도록 제어된다. 따라서 방사성 표지 된 말단(end)부터 각 분자의 첫 번째 "절단 cut" 부위까지 일련의 표지된 단편들이 생성된다.
후속 폴리아크릴아미드 겔 전기영동(polyacrylamide gel electrophoresis) 및 자동방사선 촬영(autoradiography)을 통해 ssDNA 단편을 크기별로 분리하고 DNA 서열을 코딩하는 X선 필름에서 방사선 표지된 DNA 밴드 패턴을 검출할 수 있으며, 이로부터 서열을 추론할 수 있다 (Fig-1). 네 가지 반응의 단편들은 크기 분리(size separation)를 위해 변성 아크릴아미드 겔(denaturing acrylamide gels)에서 나란히 전기영동된다. 단편을 시각화하기 위해 겔을 X선 필름에 노출시켜, 자동방사선 촬영 (autoradiography)을 실시하고, 각각 동일한 방사성 표지된 DNA 분자의 위치를 보여주는 일련의 어두운 띠 (dark bands)를 생성한다. 특정 단편들의 유무로부터, 서열을 추론할 수 있다.
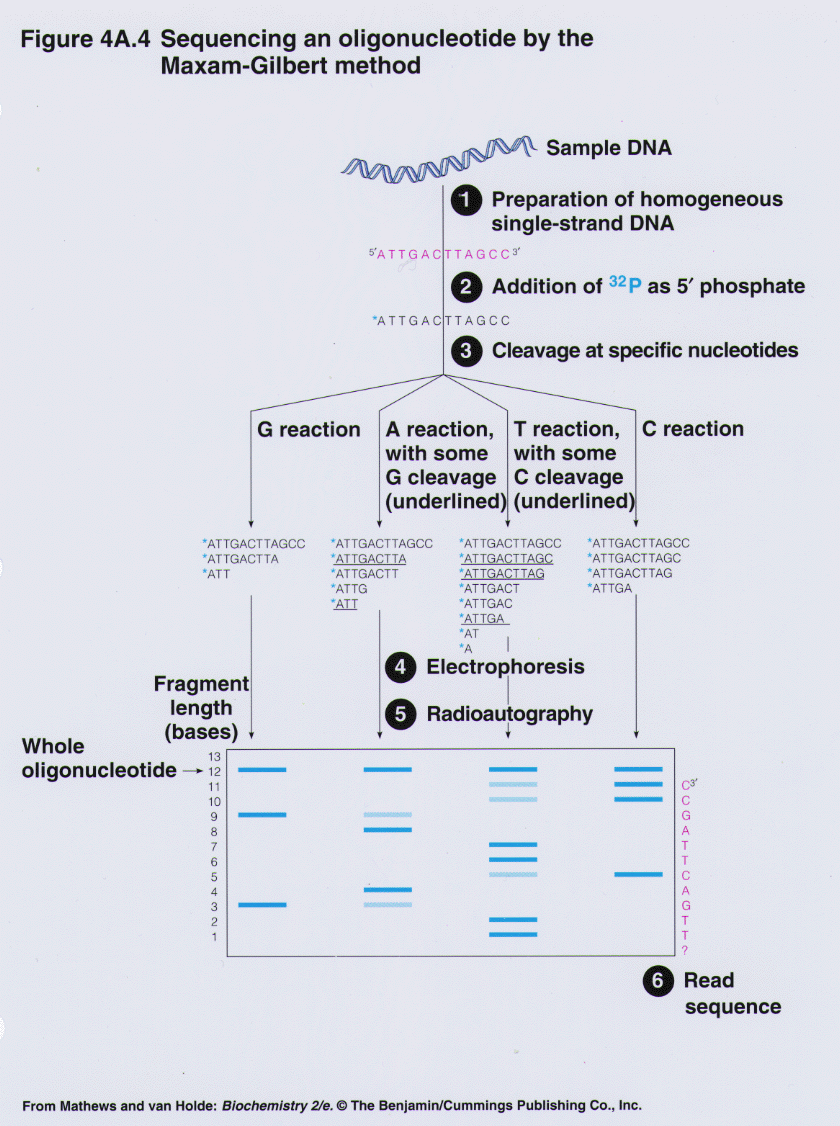
▣ Maxam-Gilbert Sequencing Steps
[1] 먼저 시퀀싱할 DNA를 상동의 한 가닥 DNA로 분리시킨다 (ssDNA).
[2] 5’ phosphate로서 방사선 물질 32P를 넣어 표시해준다 (labelling).
[3] G, A+G, C, C+T와 같은 특정 뉴클레오티드들에서 염기들을 파괴한다.
Base-specific chemical degradation을 이용해 특정 base에서 잘린 fragments를 만든다.
사용되는 화학물질들은
♣ 디메틸 황산(dimethyl sulfate) ⇒ 구아닌 (Guanine)
♣ 포름산(formic acid) ⇒ 퓨린 (purines)(Adenine + Guanine)
♣ 히드라진(hydrazine) ⇒ 피리미딘 (pyrimidines)(Cytosine +Thymine)
♣ 히드라진(hydrazine) with salt ⇒ Thymine
[4] Piperidine으로 화학처리를 한다.
화학물질들이 염기들을 파괴하면, Piperidine이 염기들이 있는 자리의 DNA 백본을 쪼갠다 (cleavage).
[4] Gel electrophoresis를 통해 분리한다.
[5] Labelled radioactive P를 이용해 autoradiography를 시행한다.

Sanger et al.은 상대적 용이성과 신뢰성으로 인해 향후 30년 동안 가장 인기 있고 널리 사용된 효소 합성 (enzymatic synthesis)에 의한 DNA 시퀀싱 어푸로치 (Sanger et al., 1977)을 개발했다. Sanger 방법론은 디데옥시 터미네이터(dideoxy terminators)를 사용하여 25 bases (b)-long reads를 생성했으며, 이후 80b로 확장되었다. 프레데릭 생어는 1980년 노벨화학상을 수상했으며, 이로서 노벨상을 두번이나 타는 위업을 남겼다.
처음에는 방사성 동위원소(radioisotopes)를 포함한 독성 화합물을 사용했지만, 나중에는 형광 디데옥시뉴클레오티드(fluorescent dideoxynucleotide) 염료(dyes)와 자동 검출(automated detection)을 사용하여 최적화했다. 이러한 개선으로 처리량과 정확도(accuracy)가 향상되어 다른 곳에서 설명한 것처럼 약 1000 b의 reads가 가능해졌다 (Lario et al., 1997).
간단히 말하면,
⊙ dsDNA (예: PCR로부터의 플라스미드(plasmid) 또는 앰플리콘(amplicon))는 변성되고(denatured),
⊙ 특정 프라이머(specific primer)는
특정 양의 디데옥시 뉴클레오티드 (dideoxy nucleotides) (ddNTP; ddATP, ddCTP, ddGTP & ddTTP)와
자연적인 것들 (dNTP; dATP, dCTP, dGTP & dTTP)을 포함하는 반응 혼합물(mix)에서
DNA 중합효소(polymerase)로 어닐링 및 확장된다.
전자는 그 당의 30번 위치에 수산기(hydroxyl)(OH) 대신 수소(hydrogen)(H)를 갖고 있기 때문에, 합성되는 ssDNA에 일단 통합되면 후속 중합을 차단한다. 따라서 이 방법론은 디데옥시 터미네이터 서열분석(dideoxy-terminator sequencing)으로 알려져 있으며, 어닐링된 프라이머가 DNA 중합효소에 의해 확장된 후 각각의 모든 뉴클레오티드 위치에서 종결된 ssDNA 분자의 확률론적 집단을 효과적으로 생성한다.
종결된 ssDNA 분자는 PAGE (PolyAcrylamide Gel Electrophoresis) 또는 CE (Capillary Electrophoresis)에 의해 분리되고 형광이 검출되어 30-말단에서 터미네이터를 식별한다. 수집된 신호를 통해 인공 색소로 DNA 서열을 나타내는 가상 전기영동도(electropherogram) 또는 전기형광도(electrofluorogram)를 생성할 수 있다 (Figure 2).
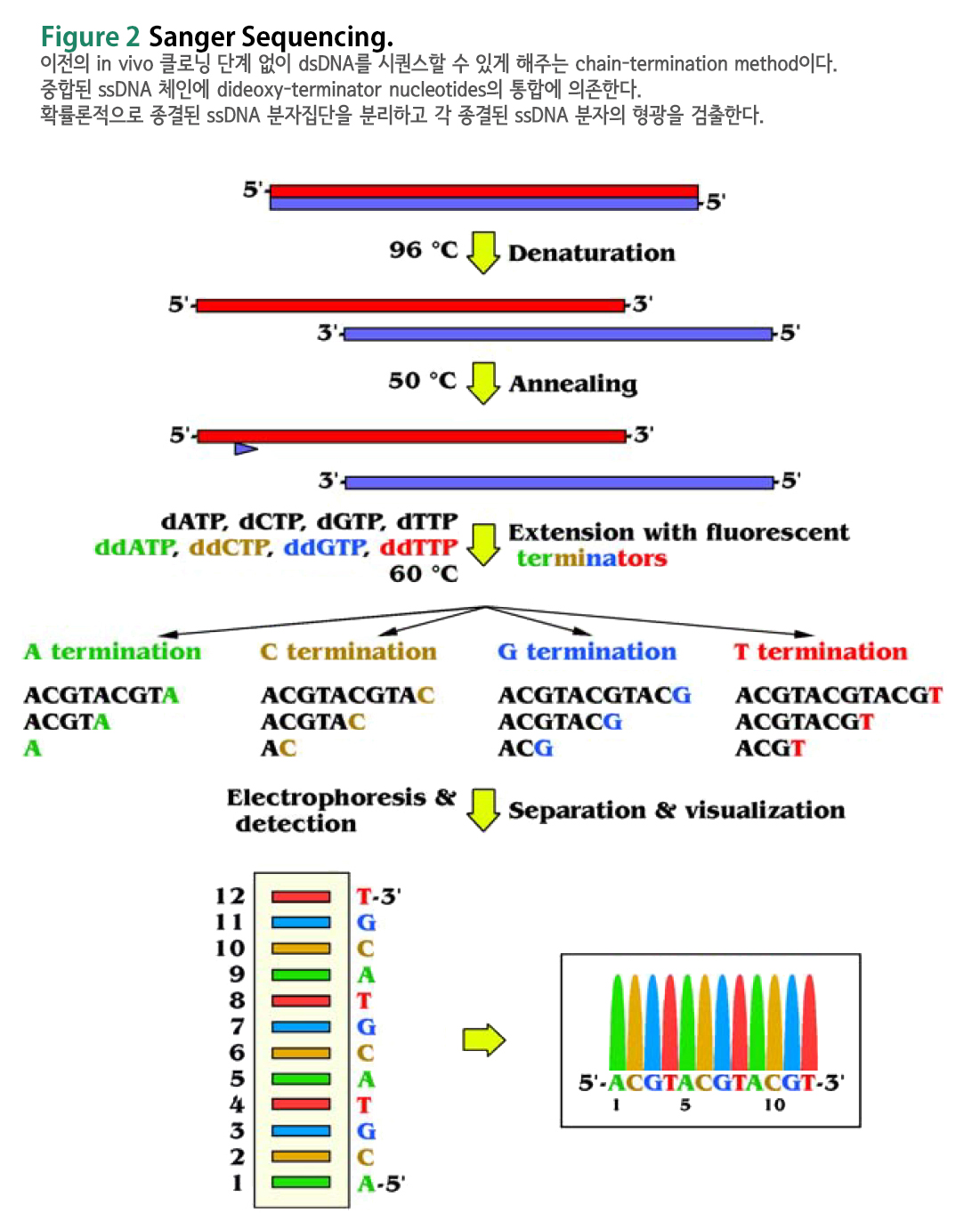
▣ Sanger Dideoxy-Terminator Sequencing Steps
ddNTP (2’,3’-dideoxynucleotide triphosphate)를 이용해 특정 base로 끝나는 fragments를 만든다.
[1] Single stranded DNA template와 labelled primer를 마련한다.
[2] 한 tube에 DNA template, Primer, DNA polymerase, 네 가지 dNTPs (A, T, G, C)와 한 종류의 ddNTP를 넣어준다.
[3] ddNTP는 3’ Oh group이 없기 때문에 phosphodiester bond를 형성하지 못해
DNA pol이 ddNTP를 붙이면 elongation이 멈춘다.
[4] 각각 ddATP, ddTTP, ddGTP, ddCTP로 끝나는 fragments가 만들어지고
이를 denaturing시켜
gel electrophoresis로 fragments 길이에 따라 분리한다.
[5] 이후, ddNTP에 각각 다른 fluorescent tag를 붙여
capillary-based, semi-automated Sanger’’s method로 발전했다.
1세대 DNA 시퀀싱 방법론들, 특히 Sanger terminator sequencing은 엽록체 및 미토콘드리아와 같은 소규모 프로젝트에 더 적합했지만 (Besnard et al., 2011) 결국 규모가 확대되었다. 동물과 식물을 포함한 고등 유기체의 큰 유전자는 물론 복잡한 게놈까지 서열 분석한다.
그래도 그런 접근은 시퀀싱 장비, 시약 및 인력에 대한 대규모 투자와 오랜 기간이 필요하다. 예로서, 반수체 인간 게놈 (haploid human genome)의 최초의 시퀀스 (30억 개의 염기쌍, 즉 3 Gigabase pairs 또는 Gbp)가, 비록 300만 달러의 비용이 들고 13년(1990~2003)이 필요하기는 했지만, 이 기술을 사용하여 완성되었다.
게다가 주목할 만한 점은 Sanger 방법론은 일반적으로 ancient DNA (aDNA)의 게놈 서열 분석에 적합하지 않다는 것을 나타낸다. 왜냐하면 고고학 샘플은 전형적으로 물리적(예: 작은) 및 화학적으로 분해된 DNA (예: 무염기 부위(abasic sites) 및 互變異性 변화(tautomeric changes))에 의해 방해되기 때문이다. 이러한 상황은 새로운 DNA 서열분석 전략 (Dorado et al., 2008)의 개발로 바뀌었다.

▣ Sanger Dideoxy-Terminator Sequencing : Principles
DNA, 즉 디옥시리보핵산 (deoxyribonucleic acid)을 합성하는데 필요한 nucleotide를
⇒ 디옥시뉴클레오시드 삼인산 (deoxynucleoside triphosphate), 줄여서 dNTP라고 부른다.
dNTP는 아래 그림처럼 5개의 탄소로 이루어진 디옥시리보오스 (deoxyribose)를 중심으로, 1번 탄소에는 염기가, 5번 탄소에는 삼인산기가 결합된 구조를 가지고 있으며, 갖고 있는 염기의 종류에 따라 dATP (아데닌), dGTP (구아닌), dCTP (시토신), dTTP (티민)으로 다시 분류할 수 있다.
dNTP에서 3번 위치에 있는 하이드록시기 (hydroxyl group, -OH)와 5번 위치에 있는 삼인산기는 DNA 합성 과정에서 매우 중요한 역할을 한다.
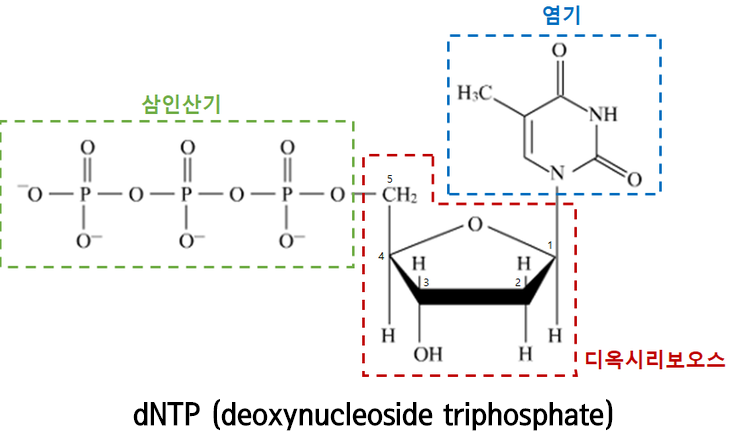
이 두 작용기는 갖고 있는 에너지가 매우 커서 주변의 다른 분자들과 반응하려는 반응성이 높다. 이러한 높은 반응성으로 인해 동일한 dNTP가 계속해서 추가적으로 결합하는 것이다.
일반적으로 DNA의 합성은 dNTP의 3’-하이드록시기에 새로운 dNTP의 5’-인산기가 결합하여 이루어진다.
이러한 과정은 5’ 위치의 삼인산기는 2개의 인산기를 잃고 1인산기 (monophosphate group)가 된다.
반응성이 높은 두 작용기가 서로 결합하여 에너지가 낮은 안정한 상태로 되려는 성질을 이용하는 것이다.
또한 이러한 반응으로 인해 DNA는 디옥시리보오스의 탄소 번호를 기준으로 항상 5번 탄소에서 3번 탄소 방향으로 합성이 일어난다.
프레데릭 생어는 이러한 DNA의 합성 과정에서 각 단계별로 사용된 dNTP의 종류를 판별할 수 있다면 결과적으로 염기서열 분석을 할 수 있다는 점을 이용했다.
예를 들어, DNA가 합성될 때, 순서대로 dATP, dATP, dTTP, dGTP가 사용되었다면, 그 DNA 분자의 염기서열은 “AATG”가 될 것이다.
문제는 DNA의 합성을 각 dNTP가 사용하는 순간순간에 멈출 방법이 필요했다는 것이다. DNA의 합성 속도는 매우 빨라서, 우리가 직접 어떤 dNTP가 사용되었는지 관찰할 방법이 없었다.
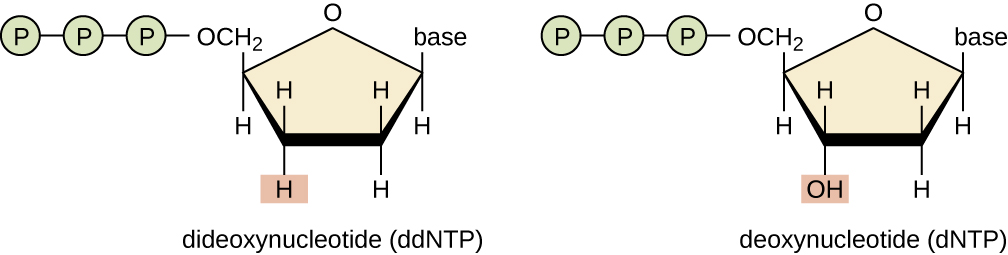
생어는 이러한 문제를 해결하기 위해 디디옥시뉴클레오시드 삼인산 (dideoxynuleoside triphosphate), 줄여서 ddNTP이라는 물질을 사용하게 된다.
ddNTP는 3번 탄소에는 OH 대신 H가 위치하고 있기 때문에, 다음 순서의 5’-인산기와 반응하는 것이 불가능하여, 더이상의 합성의 진행이 안된다. 즉, 연쇄반응이 정지되어 버리는 것이다 (chain termination).
당연히 DNA 합성 과정에 ddNTP만을 넣어주게 되면 DNA는 합성이 되지 않을 것이다.
그러나, DNA와 dNTP, 그리고 소량의 ddNTP를 같이 섞어주게 되면 어떻게 될까?
어떤 DNA 분자는 dNTP와 결합하여 계속 길이가 길어질 것이고, 어떤 DNA 분자는 ddNTP를 만나 반응이 정지되어버릴 것이다. 결국 똑같은 DNA를 주형 삼아 합성을 시작하더라도, 그 결과 합성된 DNA의 길이는 모두 다르게 될 것이다. 예를 들어, DNA 합성 과정에 소량의 ddGTP를 함께 넣어주면, 아래 그림과 같이 G가 합성되는 부분에서 일부 DNA의 연쇄반응이 정지되고, 다양한 길이의 DNA 가닥이 만들어지게 된다.
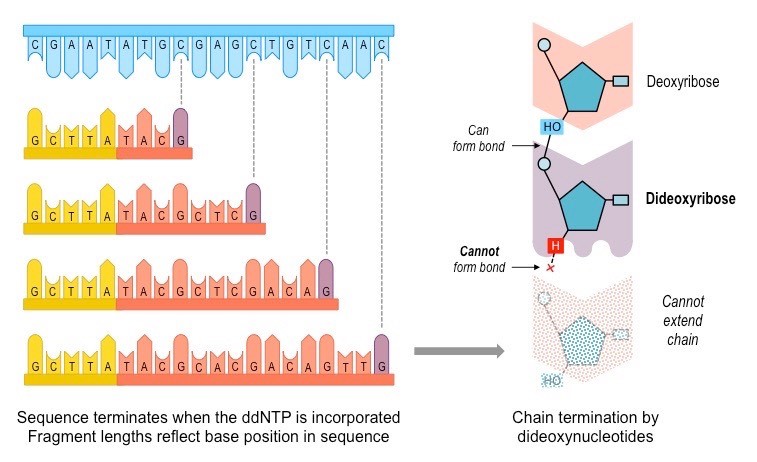
이러한 현상을 이용해, 생어는 하나의 DNA 주형의 합성 과정에서 4종류의 각기 다른 ddNTP를 이용해 반응시킨 후, 이를 각각 겔 전기영동하여 만들어진 DNA 가닥의 크기를 보았다. 이렇게 DNA 가닥을 크기별로 구분하면 결과적으로 DNA의 염기서열을 알 수 있게 된다.
다만 이런 방법으로 염기서열 분석을 진행하려면 네 종류의 ddNTP를 이용해 각각 반응을 진행해야 한다는 불편함이 있다.
이러한 불편함을 극복하기 위한 기술 개량이 지속적으로 이루어졌다.
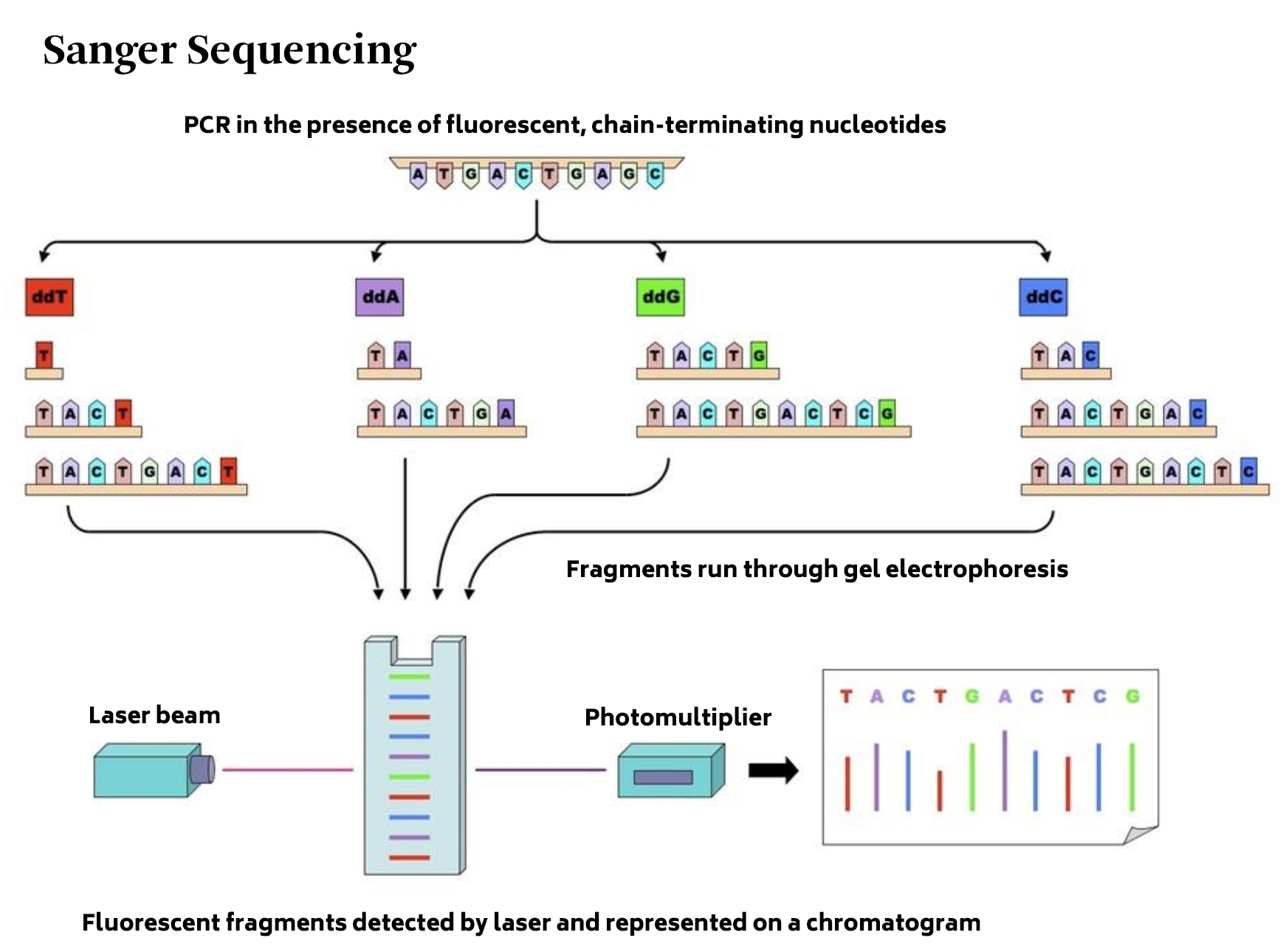
그 결과 현대에는 네 종류의 ddNTP에 각각 다른 방사능 동위원소 32P 또는 형광물질을 부착하여, 한 번에 DNA 합성을 진행하고, 이를 전기영동하면서 감지기로 표지를 감지, 한 번의 반응으로 한 DNA 가닥의 염기서열을 분석할 수 있게 되었다.
또한 한 번에 대량의 DNA 분자를 분석할 수 있도록 최근에는 완전 자동화된 기계가 널리 보급되어 쓰이고 있다.
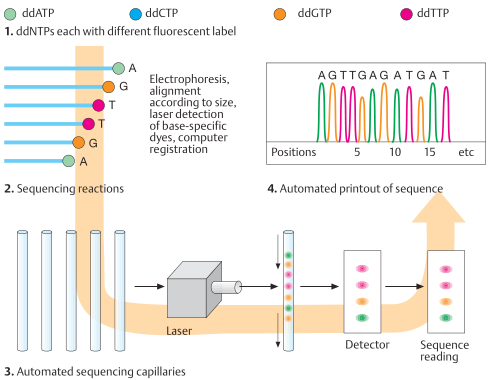
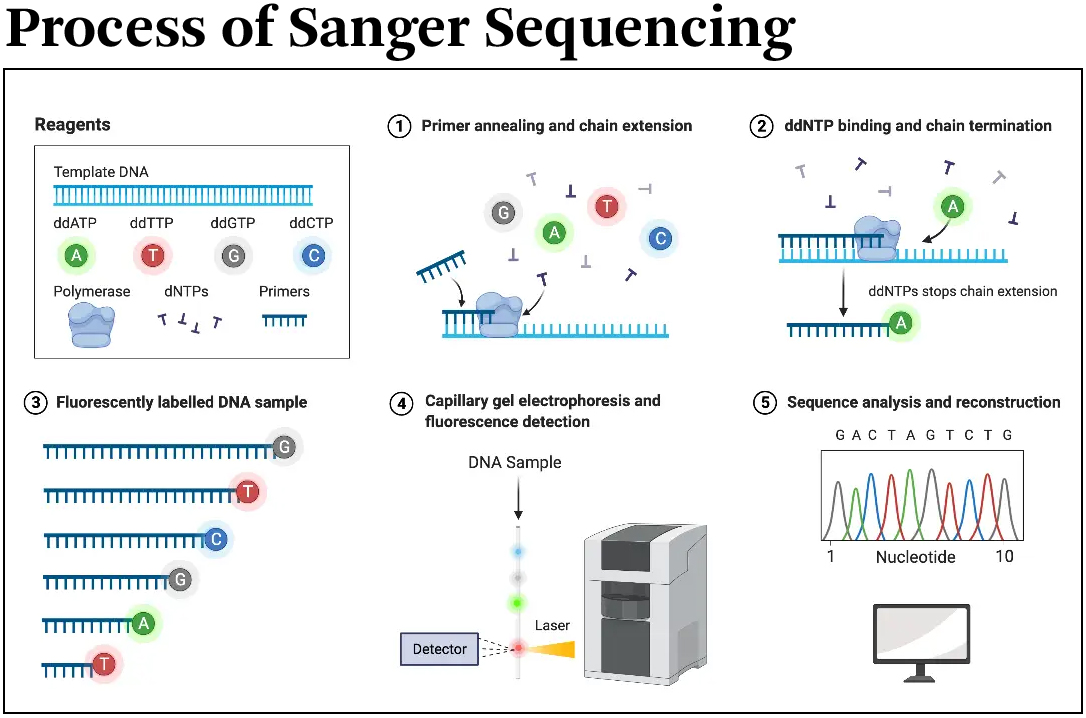
▣ Sanger Sequencing Method : Principle & Steps
DNA (deoxyribonucleic acid) sequencing은 뉴클레이티드들의 정확한 서열(sequence)를 식별하는 프로세스이다: 게놈 또는 DNA 분자에 있는 Adenine (A), Guanine (G), Cytosine (C), and Thymine (T).
그 시퀀스를 결정하기 위해서, 첫번째 DNA sequencing method, the “chain termination method” 또는 Sanger sequencing은 방사능 표지된 부분적 분해된 단편들을 사용하여, Frederick Sanger에 의해서 1997년에 개발되었다.
이 방법으로 Frederick Sanger와 그의 팀은 phiX174 바이러스의 최초의 완전한 게놈을 시퀀싱할 수 있었다.
Sanger sequencing은 in vitro DNA replication (복제) 동안에 사슬 종결 올리고뉴클레이티드들(oligonucleotides)의 통합이 프라이머들로 발생하는 시퀀싱 방법이다.
생어 시퀀싱의 필수 구성요소들은 다음과 같다.
⊙ Single-stranded DNA molecules,
⊙ DNA polymerase (중합효소),
⊙ 4가지 디옥시리보뉴클레오티드 삼인산염
(deoxyribonucleotide triphosphates, dNTPs; dATP, dCTP, dGTP, dTTP), 그리고
⊙ 여러 형광물질 마커들로 표시된 디데옥시리보뉴클레오티드 삼인산염
(dideoxyribonucleotides triphosphates (ddNTPs; ddATP, ddCTP, ddGTP, ddTTP).
Sanger Sequencing method는 deoxynucleotide triphosphate, 그리고 신장(elongation)을 종결하는, 여러 다른 형광물질 마커로 표시된 dideoxynucleotide triphosphates를 더해줌으로써 한 가닥의 DNA 템플릿에 상보적인 DNA를 합성한다. 이 표시들에 따라서, 서열이 확인된다.
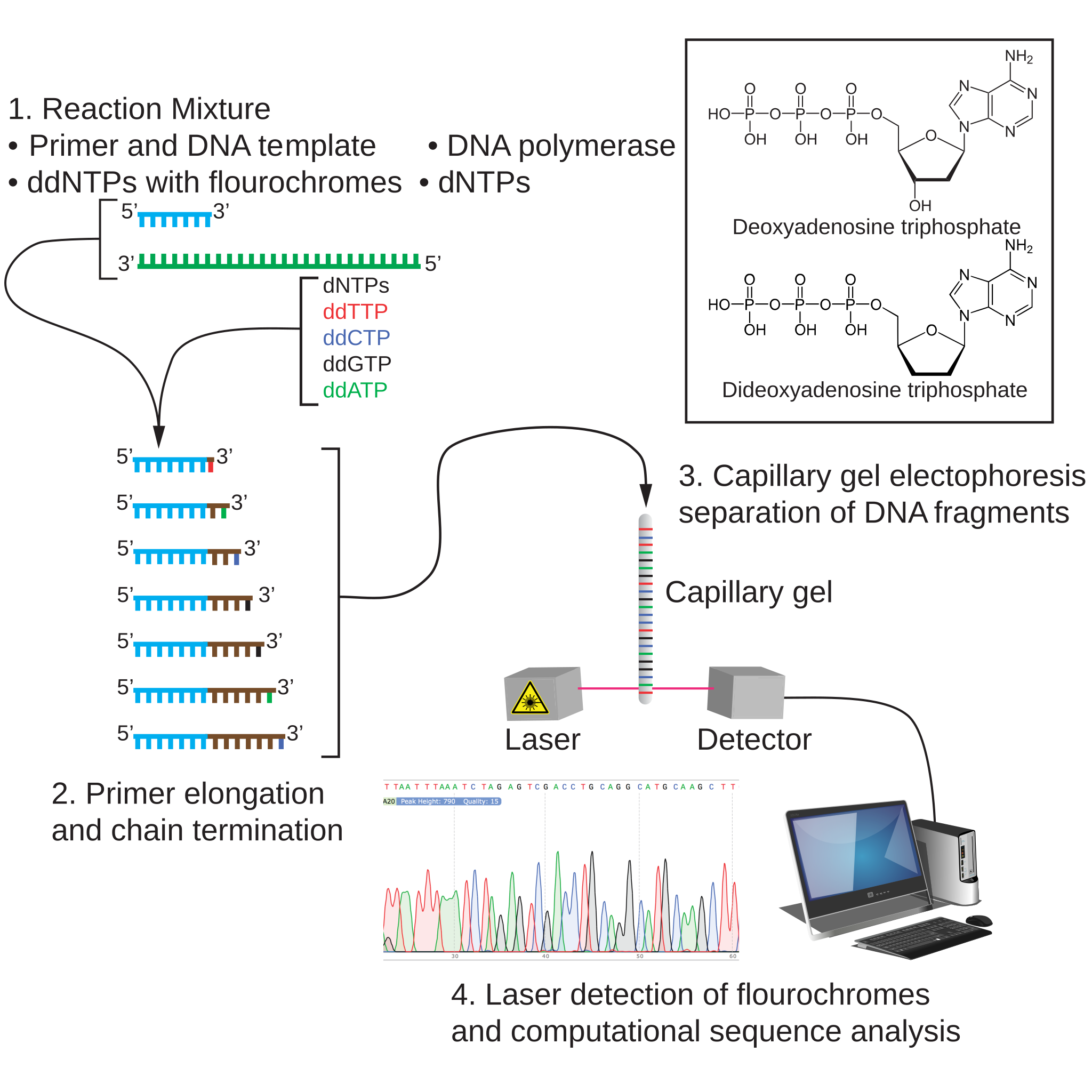
▣▣▣ Sanger Sequencing Method : Components of Sequencing ▣▣▣
▣ DNA template
시퀀싱의 시작 물질은 서열분석되어야 할 싱글 가닥 DNA 템플릿(a single-stranded DNA template)이다.
전통적인 방법에서는, single-stranded DNA를 얻기 위해 벡터를 사용하거나 또는 alkali method 또는 boiling method로 double-stranded DNA를 변성(denaturing )시킨다. 그러나, 요즘에는, polymerase chain reaction (PCR, 중합효소연쇄반응) method가 single-stranded DNA를 얻는데 도움이 된다.
▣ Primer
프라이머는 DNA 템플릿들의 짧은 시퀀스에 상보적인 짧은 올리고뉴클레오티드이며, 그것으로 어닐(anneal)한다 (가열처리 후 식힘). 시퀀스될 템플릿 분자의 영역이 프라이머가 하는 핵심 역할이다.
▣ DNA polymerase enzyme
중합효소는 프라이머를 연장하고, 그 템플릿에 상보적인 뉴클레오티드를 더해준다.
이 중합효소는5ʹ- 3ʹ 또는 3ʹ- 5ʹ 로부터 뉴클레오티드를 분해하여 그 시퀀스 결정의 정확도에 영향을 미칠 수도 있으므로, exonuclease activity (엑소뉴클레아제, 핵산분해효소) 작용이 부족하다.
▣ Nucleotide triphosphate (뉴클레오티드 삼인산염):
이 방법에는 두 가지 다른 유형의 뉴클레오티드, 즉 데옥시리보뉴클레오티드 삼인산염과 디데옥시리보뉴클레오티드 삼인산염이 사용된다.
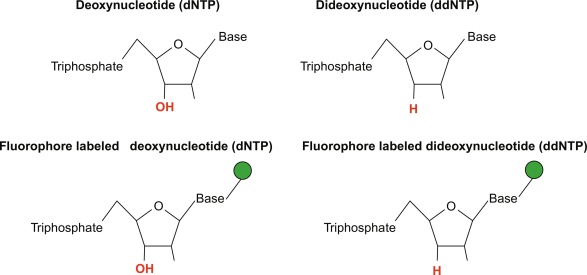
- Deoxyribonucleotide triphosphates (디옥시리보뉴클레오티드 삼인산):
이는 전형적인 뉴클레오티드 삼인산으로, 질소 염기들 (A, T, G, C), 3' 탄소에 수산기(hydroxyl group), 5' 탄소에 인산기(phosphate group)를 각각 갖는 리보스 당(ribose sugar)으로 구성되어 있으며, 이는 각 디옥시리보뉴클레오티드 삼인산 사이에 인산디에스테르 결합(phosphodiester bond)을 형성하는 데 도움이 된다. - Dideoxyribonucleotide triphosphates (디데옥시리보뉴클레오티드 삼인산):
리보스 당의 3' 탄소에 수산기가 없도록 변형된 뉴클레오티드 삼인산(nucleotide triphosphates)이다.
따라서 포스포디에스테르 결합이 다음 들어오는 뉴클레오티드와 함께 형성되지 않기 때문에
이들은 3’-end chain terminators로 사용된다.
▣▣▣ Sanger Sequencing Method : Steps ▣▣▣
▣ Elongation and chain termination
시퀀싱할 DNA 템플릿은 dideoxynucleotide triphosphate도 포함되어 Polymerase Chain Reaction (PCR)을 거친다.
dNTP, DNA 중합효소(polymerase), 프라이머 및 템플릿은 모두 일반적인 PCR 반응에 포함된다.
프라이머는 템플릿 DNA에 어닐링하고, DNA 중합효소는 dNTP 또는 ddNTP를 무작위로 추가하여 프라이머를 확장하지만, ddNTP가 통합되면 반응이 종료된다. 이러한 데옥시뉴클레오타이드는 디데옥시뉴클레오타이드보다 크기 때문에 종료는 프라이머 영역 근처에서 발생하지 않는다. 예를 들어, dATP가 연장 중에 결합하면 연장이 중단되고 시퀀스는 A로 읽힌다. 마찬가지로 ddGTP, ddTTP 또는 ddCTP가 추가되면 시퀀스는 각각 G, T 또는 C로 읽힌다.
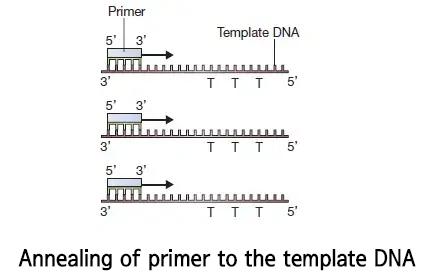
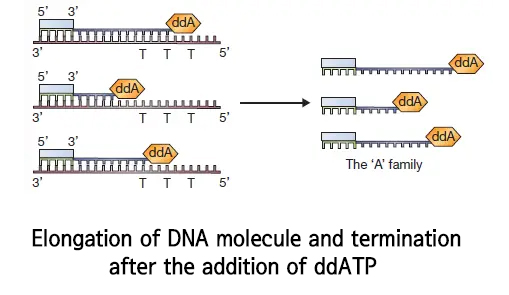
전통적인 방법에서는, 전형적인 PCR 혼합물을 담은 4개의 튜브들에서 반응을 수행한다.
아직, 각 튜브에는 방사능 표지된 디데옥시뉴클레오티드 삼인산 (ddATP, ddCTP, ddTTP, or ddGTP)을 포함하고 있다.
한편, 요즘에는 각기 다른 형광염료로 표지된 ddNTPs를 하나의 큐브에 넣어 반응이 수행된다.
▣ Electrophoresis and Sequence Identification
증폭(amplification) 후에 전기영동(electrophoresis)이 수행된다.
혼합물은 polyacrylamide gel (전통적인 방법)이나, 또는 모세관 겔 시스템(capillary gel system) 튜브에 넣는다.
이 분자들은 그 길이에 따라 분리된다; 각각은 끝 부분에 디데옥시뉴클레이티드를 가지고 있다.
전통적인 방법에서는, 그 결과물들은 4가지 분리된 레인들에서 폴리아크릴아미드 겔 전기영동을 통해 분리되어지고, 아래 그림에서 나타난 바와 같이 그 분자량에 따라 스코어링된다.
그들의 질량(masses)에 따라서, 그리고 방사능 표지된 ddNTPs에 따라서 염기들이 식별된다.
그들의 시퀀스들이 그림 중 아래부분의 왼쪽면에 나와 있다. 그 식별은 spectrophotometer의 도움으로 이뤄진다.
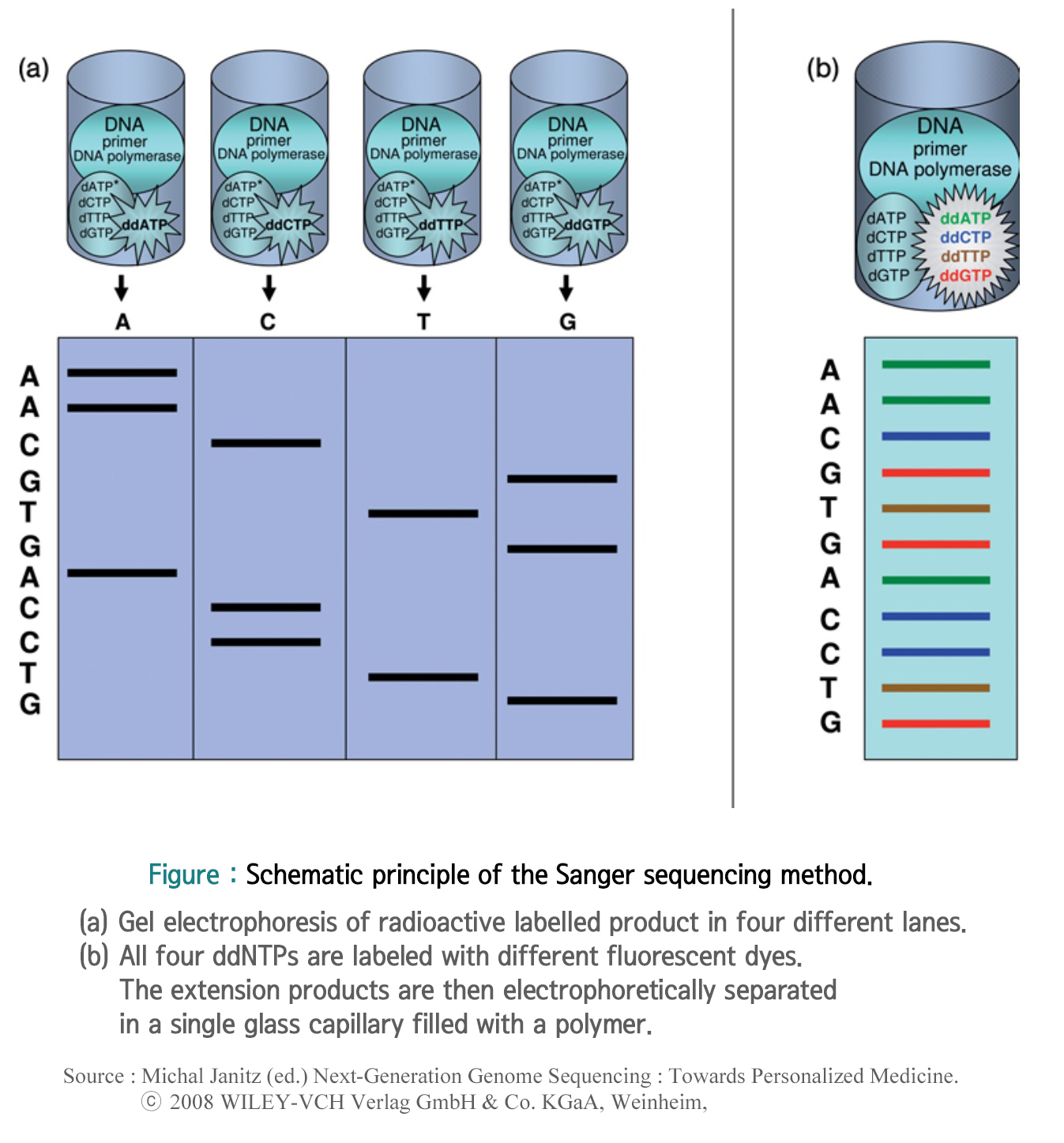
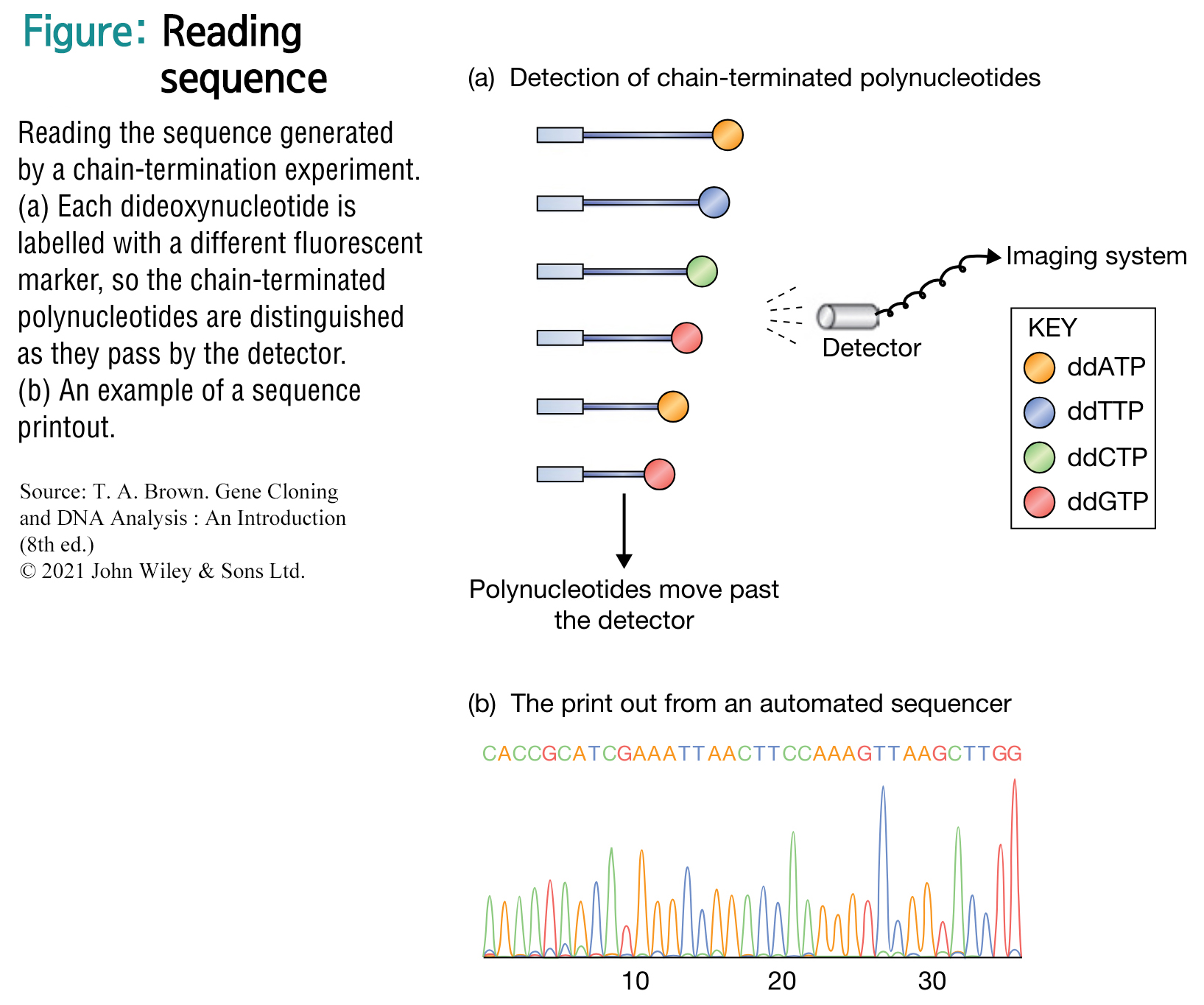
▣ Advantages of Sanger Sequencing
- 동일한 패밀리의 변이체들을 테스트하는 데 더 구체적이다.
- 이 방법은 하나 또는 소수의 관심 시퀀스에 대해 검증되므로 광범위한 검증이 필요하지 않다.
- 계산 도구에 대한 의존도가 줄어든다.
- 단일 샘플에 대해 비용 효율적이다.
▣ Limitations of Sanger Sequencing
- 이는 약 300-1kb 염기의 짧은 DNA 단편만을 서열화한다.
- 서로 다른 유전자를 동시에 검출할 수 없다.
- 입력으로 더 많은 양의 DNA가 필요하다.
- 프라이머가 처음 15~40개의 염기에 결합하기 때문에, 이 영역의 서열 품질이 좋지 않은 경우가 많다.
- 시간이 많이 걸리는 방법이다.
- 전통적인 방법을 사용하는 경우 클로닝 벡터 서열이 최종 서열에 존재할 수 있다.

Sanger method는 직관적으로 밑에서부터 바로 읽어가며, Maxam-Gilbert method는 dual bank인지 아닌지 확인하며 읽는다. 예를 들어 Lane for A와 lane for A+G 둘 다 band를 가지고 있다면 G, lane for A+G에서만 나타났다면 A라고 읽는다.
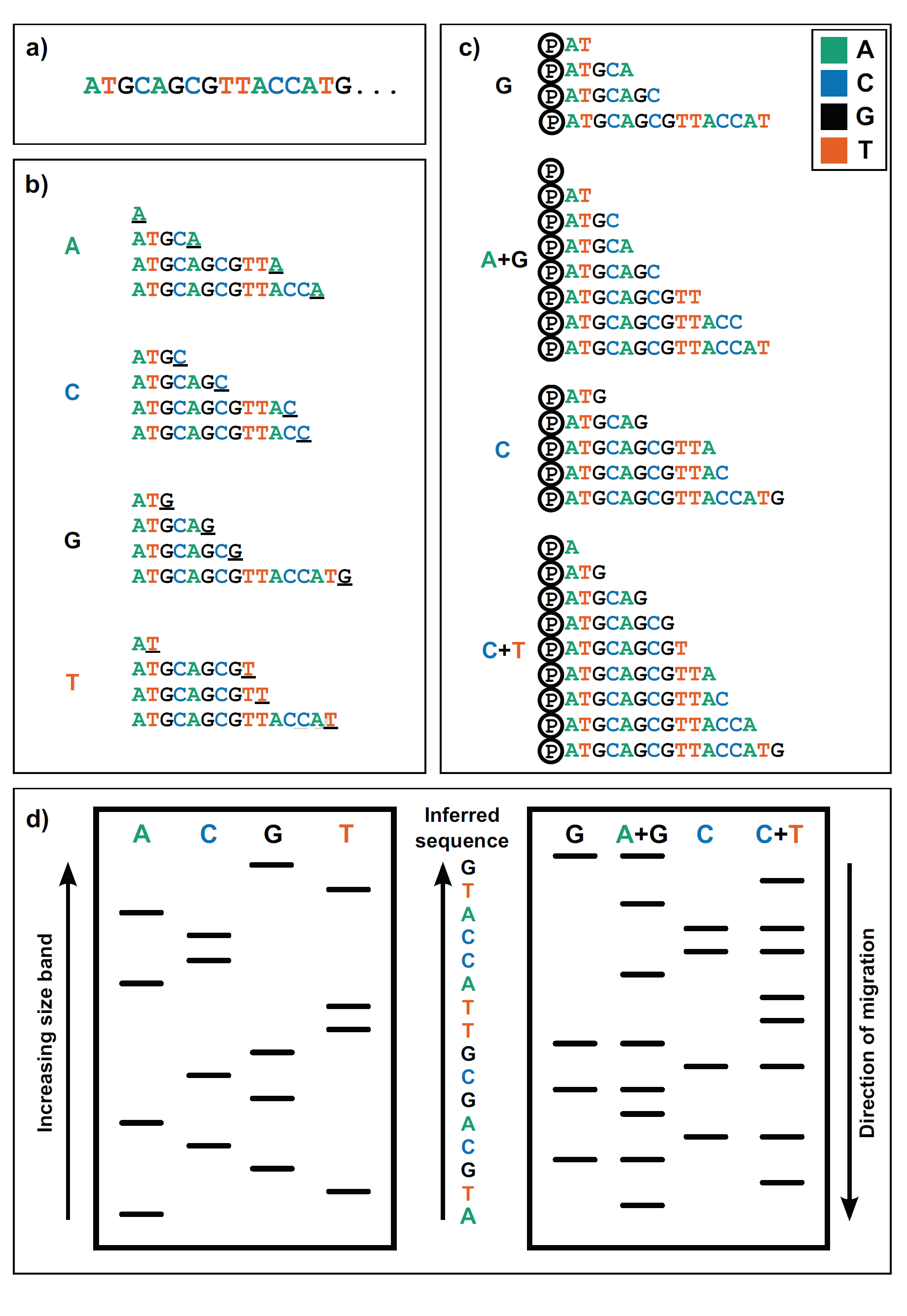
Figure : First-generation DNA sequencing technologies.
시퀀싱할 예시 DNA (a)는 Sanger sequencing (b) 또는 Maxam-Gilbert sequencing (c)을 거치는 것으로 그려져 있다.
(b): Sanger's ‘chain-termination’ sequencing. 일단 통합되면 추가 확장을 방지하는 특정 유형의 방사성-표지된 또는 형광-표지된 ddNTP 뉴클레오티드는 DNA 중합 반응에 저 농도로 포함된다 (5’ 시퀀스에서 primed됨, 그림에서 나와 있지는 않음). 따라서 네 가지 반응 각각에서 ddNTP가 해당 염기의 특정 인스턴스 (밑줄 친 3’ terminal 문자)에 무작위로 통합됨에 따라 3’ 절단 (truncations)을 사용하여 서열 단편이 생성된다.
(c): Maxam-Gilbert의 ‘chemical sequencing’ method.
일반적으로 DNA의 5' 인산 부분(여기서는 Ⓟ로 표시)에 방사성 P32를 포함시켜 DNA를 표지해야 한다. 그런 다음 서로 다른 화학 처리를 사용하여 소량의 DNA 부위에서 염기를 선택적으로 제거한다. 히드라진 (Hydrazine)은 pyrimidines (cytosine과 thymine)에서 염기를 제거하는 반면, 농도가 높은 소금이 추가된 히드라진은 cytosine에서만 염기를 제거할 수 있다. 그런 다음 산(acid)을 사용하여 purines (adenine 및 guanine)에서 염기를 제거할 수 있으며, 디메틸 황산(dimethyl sulfate)은 guanines를 공격하는 데 사용된다 (아데닌도 훨씬 덜 영향을 받긴 하지만). 그런 다음 피페리딘(Piperidine)을 사용하여 무염기 부위(abasic site)에서 포스포디에스테르 백본 (phophodiester backbone)을 쪼개서 (cleave) 다양한 길이의 단편들을 생성한다.
(d): 두 방법 중 하나에서 생성된 단편들은 고해상도 폴리아크릴아미드 겔(polyacrylamide gel)에서 전기 영동을 통해 시각화될 수 있다. 그런 다음 짧은 DNA 단편이 가장 빠르게 이동하므로 겔을 '위로' 읽어서 서열을 추론한다. Sanger sequencing (왼쪽)에서는 주어진 부위에 밴드가 존재하는 레인을 찾아 서열을 추론한다. ddNTP로 표시된 3' 종결 부분이 해당 위치의 염기에 해당하기 때문이다. Maxam-Gilbert sequencing (오른쪽)에서는 약간의 추가 논리적 단계가 필요하다. Ts와 As는 각각 피리미딘 또는 퓨린 레인의 밴드에서 직접 추론할 수 있다. G와 C는 각각 G 및 A + G 레인 또는 C 및 C + T 레인에 듀얼 밴드들의 존재로 표시된다.
'Coffee Genetics' 카테고리의 다른 글
| Illumina Sequencing Technology - 2세대 DNA 시퀀싱 (22) | 2024.07.11 |
|---|---|
| Roche 454 Life Sciences Emulsion-PCR Sequencing : 2세대 DNA 시퀀싱 (0) | 2024.07.06 |
| 식물 세포, 핵, DNA, 유전자, 게놈 (1) | 2024.06.29 |
| 아라비카 커피의 기원과 多樣化 歷史 (Coffea arabica diversification history) (0) | 2024.06.15 |
| 아라비카 커피 기원의 싱글 다배체화 사건이 극히 낮은 유전적 변이의 원인 (0) | 2024.06.05 |




댓글