
전 세계인들이 가장 즐겨 마시는 아라비카 커피의 게놈 연구를 통한 아라비카 커피의 탄생 비밀과 기원 스토리를 과학적으로 밝힌 엄청난 논문이 국제 학술지 『'Nature Genetics' 네이처 유전학』 2024년 4월호의 표지를 장식하며 발표되었다. 에티오피아 고원이 원산지인 아라비카 커피는 15세기 예멘에서 재배되었다. 아시아와 아메리카에 도착하는 모험적인 상황에 대해 자세히 조사하지 않고도 새로운 재배지역에 대해 초기 개체군은 매우 적은 수의 개체 또는 심지어 몇 개의 종자에서 유래했다는 점에 주목할만한 가치가 있다. 따라서 전문가들은 현재 전 세계 아라비카 개체수의 유전적 기반이 제한적이라고 말한다 (유전적 다양성이 낮다는 의미). 아라비카 커피 나무는 50만 년 전 두 야생 종인 Coffea canephora (Robusta)와 C. eugenioides 사이에서 자발적인 교배가 이루어진 식물의 후손이다.
두 가지 역사적 혈통인 티피카(Typica)와 버번(Bourbon) 및 그 파생 품종을 통해 재배되는 아라비카 커피의 다양성은 매우 낮기 때문에, 생산이 기후나 위생 위험에 특히 민감하다. 현대 품종 개량에 대한 전문가들을 계몽하기 위해 18개국의 과학자들이 IRD (Institut de Recherche pour le Développement)와 Nestlé의 주도 하에 협력하여 세 가지 게놈(아라비카 및 그 야생 부모)의 서열 분석에 착수했다.
그들이 유전적 보따리 속에서 버번 포인투, 모카, 블루 마운틴과 같은 가장 잘 알려진 12가지 품종과 그 친척을 낳은 에피소드를 밝히는 데 10년이 걸렸다. UMR (Unité Mixte de Research) DIADE (Diversity-Adaptation-Development of Plants)의 유전학자인 Valérie Poncet은 "이것은 우간다, 브라질, 콜롬비아와의 오랜 파트너십이 없었다면 불가능했을 것입니다"라고 강조한다. "따라서 C. eugenioides의 서열화된 개체는 NARO의 파트너와 함께 연구한 두 종인 C. canephora 와 여전히 공존하는 우간다 숲에 살고 있습니다. C. arabica의 경우, 다음을 제공한 것은 런던 자연사 박물관이었습니다. 린네가 종을 정의할 수 있게 해준 식물표본관 표본입니다.”
아라비카의 경우 가장 어려운 점은 부모로부터 물려받은 두 서브 게놈의 서열을 분석하고 이를 두 부모 종의 현재 게놈과 비교하여 구별하는 것이었다. "이것은 IRD의 살아있는 컬렉션에서 예외적인 개체들에 접근함으로써 가능해졌습니다" 라고 조인트 연구 유닛 DIADE의 유전학자이자 또 다른 공동 저자인 Romain Guyot는 덧붙인다. 과학자들은 "두 서브 게놈 중 어느 것도 발현 측면에서 다른 서브 게놈을 지배하지 않습니다. C. arabica는 두 부모 간의 완벽한 협력의 결과입니다" 라고 지적한다. 맛의 품질은 이러한 균형에 기인한다고 여겨진다.
아라비카 커피가 이렇게 성공한 것은, 씁쓸하고 묵직한 로부스타 커피에 비해 섬세한 풍미와 비교할 수 없는 훌륭한 맛이 있기 때문이다. 따라서 연구의 저자들은 이러한 인기 있는 특성을 담당하는 유전자군을 조사했다. 그들은 주로 브라질 농업 연구 회사인 EMBRAPA에서 야생 및 재배 다양성을 대표하는 40개의 개체를 서열 분석했다.
최첨단 DNA 시퀀싱 기술과 데이터 과학을 활용하여 아라비카 종의 새로운 레퍼런스 게놈을 완성하고, 아라비카 품종 39개를 시퀀싱하고, 카네포라 품종, 유게니오이데스 종의 커피 유전체를 분석했다. 공동 교신저자 중의 한명인 Nestlé Research의 유전체학 수석 전문가인 Patrick Descombes는 "아라비카 커피에 대한 다른 공개 참고자료가 존재하지만, 우리 팀의 작업 품질은 매우 높습니다. 우리는 현재까지 가장 진보되고 완전하며 연속적인 아라비카 레퍼런스 게놈을 생성하기 위해 long- and short-read high throughput DNA sequencing를 포함한 최첨단 유전체학 접근 방식을 사용했습니다."라고 한다.
아라비카 커피의 최초 등장 시기를 확인했다. 약 61만년~100만년 전에 에티오피아 숲에서 코페아 카네포라와 코페아 유게니오이데스 간의 자연적 이종간 교배 사건으로 아라비카 커피가 탄생했다는 것을 보여주었다. 아라비카 커피는 Coffea canephora 와 Coffea eugenioides 사이의 자연 교배로 형성되었으며, 이때 각 부모로부터 두 세트의 염색체를 받았다. 과학자들은 이 사건이 언제 어디서 발생했는지 정확하게 파악하는데 어려움을 겪었으며, 추정치는 10,000년에서 100만년 전까지 다양하다. 오리지날 사건에 대한 증거를 찾기 위해 연구자들은 컴퓨터 모델링 프로그램을 통해 다양한 아라비카 게놈을 실행하여 종의 기초에 대한 특징을 찾았다. 이 모델은 아라비카 역사 동안 세 번의 개체군 병목현상을 보여주며, 가장 오래된 것은 약 29,000 세대, 즉 610,000년 전에 발생했다. 이는 아라비카가 그 이전, 즉 61만년에서 100만년 전에 형성되었음을 시사한다. “즉, 아라비카를 탄생시킨 교배는 인간이 한 것이 아니었습니다.”라고 공동 교신저자이자 뉴욕 버팔로 대학의 식물 진화 생물학자인 빅터 알버트 (Victor Albert) 박사는 말한다. “이 사건이 현생인류와 커피 재배보다 앞서 있었던 것이 분명합니다.” 이 연구팀은 아라비카 커피의 게체수 급감 기간의 존재로 유전적 다양성이 대폭 감소하는 병목현상이 총 3번 있었다고 분석했다.
2만~10만년 전에는 아라비카 커피 개체수가 적은 시기가 오래 지속되었다. 이 기간은 동아프리카 지역에서의 장기적 가뭄과 서늘한 기후가 있었던 4만~7만년 전과 거의 일치했다.
약 6000년~1만5천년 전에는 아라비카 커피가 성장하기 좋은 습한 기후였기 때문에 개체수가 증가했던 것으로 확인되었다. 아라비카 커피는 수천년에 걸쳐 지구 기후변화로 개체수가 감소했다가 증가했으며, 에티오피아와 예멘에서 재배되기 시작하여 전세계로 확산했다. 공동 교신저자이자 뉴욕 버팔로 대학의 식물 진화 생물학자인 빅터 알버트 (Victor Albert) 박사는 “우리는 오늘날 살아 있는 식물의 게놈 정보를 사용하여 시간을 거슬러 올라가 아라비카의 오랜 역사를 가능한 가장 정확하게 그려냈을 뿐만 아니라 현대 재배 품종이 서로 어떻게 관련되어 있는지를 알아냈습니다.”라고 연구를 소개했다.

커피나무는 에티오피아에서 발달한 것으로 오래동안 생각되어왔으나, 연구팀이 동남 아프리카에서부터 아시아까지 뻗어있는 Great Rift Valley 주변에서 수집한 품종들은 명확한 지리적 분리를 보였다. 연구된 모든 야생 품종들은 리프트 밸리의 서쪽으로부터 유래되었고, 재배 품종들은 모두 아프리카와 예멘을 분리하는 the Bab al-Mandab (“Gait of Tears”) strait에 가장 가까운 리프트 밸리의 동쪽 지역으로부터 유래했다고 확인되었다. 이는 커피 재배가 주로 15세기경 예멘에서 시작되었을 수 있다는 증거와 일치한다. 인도의 승려 바바 부단(Baba Budan)이 1600년경 예멘으로부터 전설적인 "일곱 개의 씨앗"을 밀반출하여 인도산 아라비카 품종을 확립하고 오늘날 커피가 전 세계로 진출할 수 있는 토대를 마련한 것으로 알려져 있다.
“예멘 커피의 다양성이 현재의 모든 주요 품종의 창시자가 될 수 있는 것 같습니다.”라고 역시 공동 교신저자인 Nestlé Research의 Patrick Descombes는 제안했다. 그는 “커피는 새로운 품종을 만들기 위해 옥수수나 밀과 같이 심하게 교배된 작물이 아닙니다. 사람들은 주로 자신이 좋아하는 품종을 선택해 키웠기 때문에 오늘날 우리가 갖고 있는 품종은 아마도 오래전부터 존재했을 것입니다.”라고 설명했다.
저자들은 아라비카 커피 표준유전체로 아라비카 품종 중 하나인 티모르 종이 질병에 강한 저항력을 갖게된 이유도 설명했다. 티모르 섬에서 아라비카와 모체 중 하나인 코페아 카네포라 사이의 자연교배로 만들어진 티모르종은 질병저항성 단백질 RPP3 계열 유전자와 질병에 저항하는 기능을 조절하는 CPR1을 갖고 있는 것으로 확인되었다. 공동 교신저자로서 저자 리스트의 맨 앞에 나오는 자르코 살로야르비 (Jarkko Salojärvi) 난양공대 교수는 "이번 연구로 아라비카의 병원체 저항력을 개선할 수 있을 것"이라고 말했다.
그 동안 이론적으로 제시되고 그리고 과학적으로 점차 검증되고 더욱 밝혀져온 아라비카 커피의 기원과 진화 과정에서의 유전적 현상들에 관한 흥미로운 주제를 수년간의 노력으로 보여준 이번 논문은 경이로운 작업 결과임에 분명하고 시간을 들여 읽어볼 가치가 분명히 있다. 또한, 방법론적 측면에서, “우리는 최신 게놈 기술, 즉 고정밀 PacBio 시스템 (유전자 서열분석용)의 long reads과 Illumina (유전변이 및 생물학적 기능분석을 위한 통합시스템)의 short reads를 통한 근접 연결(proximity lignation)을 사용하여 염색체 어셈블리(chromosome assembly)를 생성했습니다. 이 조합으로 최고 품질과 완전성을 갖춘 염색체 수준의 어셈블리가 탄생했습니다.”라고 하는 Patrick Descombes의 설명에 관련된 부분들도 확인해보는 유익한 기회가 될 것으로 생각된다. 아울러 Dovetail Genomics사의 유전체학 연구 발전(게놈의 조립 및 분석)에 혁신적인 대안으로 떠오른 Hi-C 기술의 면모도 체감할 수 있는 측면도 보여주는 논문이라는 점도 주목할만하다. 이 논문의 전문은 제시된 URL에서 다운로드 받을 수 있다. https://doi.org/10.1038/s41588-024-01695-w

- School of Biological Sciences, Nanyang Technological University, Singapore, Singapore.
- Organismal and Evolutionary Biology Research Programme,
University of Helsinki, Helsinki, Finland. - Singapore Centre for Environmental Life Sciences Engineering,
Nanyang Technological University, Singapore, Singapore. - Boyce Thompson Institute, Cornell University, Ithaca, NY, USA.
- Department of Mathematics and Statistics, University of Ottawa, Ottawa, Ontario, Canada.
- Institut de Recherche pour le Développement (IRD),
Université de Montpellier, Montpellier, France. - Department of Electronics and Automation,
Universidad Autónoma de Manizales, Manizales, Colombia. - Société des Produits Nestlé SA, Nestlé Research, Tours, France.
- Institute of Biotechnology, University of Helsinki, Helsinki, Finland.
- Institute of Computing, University of Campinas, Campinas, Brazil.
- Department of Computer Science,
The Federal University of Technology – Paraná (UTFPR), Cornélio Procópio, Brazil. - Plant Sciences Unit, Flanders Research Institute for Agriculture,
Fisheries and Food (ILVO), Melle, Belgium. - Department of Biological Sciences, University at Buffalo, Buffalo, NY, USA.
- Centre d’Immunologie de Marseille-Luminy, Aix Marseille Université, Marseille, France.
- Société des Produits Nestlé SA, Nestlé Research, Lausanne, Switzerland.
- Department of Computer Science, University of Leipzig, Leipzig, Germany.
- Interdisciplinary Center for Bioinformatics, University of Leipzig, Leipzig, Germany.
- Group of Genomics and Transcriptomes in Plants,
São Paulo State University, UNESP, Rio Claro, Brazil. - Centro de Ciências Agrárias, Universidade Estadual de Londrina, Londrina, Brazil.
- Indonesian Coffee and Cocoa Research Institute (ICCRI), Jember, Indonesia.
- University of Illinois at Urbana-Champaign, Urbana, IL, USA.
- Research Unit in Plant Cellular and Molecular Biology,
University of Namur, Namur, Belgium. - Departamento de Ciencias Biológicas, Facultad de Ciencias Exactas y Naturales,
Universidad de Caldas, Manizales, Colombia. - National Agricultural Research Organization (NARO), Entebbe, Uganda.
- Biodiversité Gènes & Communautés, INRA, Bordeaux, France.
- Hortus Botanicus Amsterdam, Amsterdam, the Netherlands.
- Departamento de Biología y Geología, Universidad de Almería, Almería, Spain.
- Instituto Agronômico (IAC) Centro de Café ‘Alcides Carvalho’,
Fazenda Santa Elisa, Campinas, Brazil. - Embrapa Café/Instituto Agronômico (IAC) Centro de Café ‘Alcides Carvalho’,
Fazenda Santa Elisa, Campinas, Brazil. - Instituto de Desenvolvimento Rural do Paraná- IAPAR, Londrina, Brazil.
- Department of Plant Biotechnology and Bioinformatics,
Ghent University, Ghent, Belgium. - Queensland Alliance for Agriculture and Food Innovation,
University of Queensland, Brisbane, Queensland, Australia. - CIRAD - UMR DIADE (IRD-CIRAD-Université de Montpellier) BP 64501, Montpellier, France.
- Department of Biochemistry, Genetics and Microbiology,
University of Pretoria, Pretoria, South Africa. - College of Horticulture, Academy for Advanced Interdisciplinary Studies,
Nanjing Agricultural University, Nanjing, China. - Center for Plant Systems Biology, VIB, Ghent, Belgium.
- Embrapa Café/Inovacafé Laboratory of Molecular Genetics Campus da UFLA-MG, Lavras, Brazil.
- Italian National Agency for New Technologies,
Energy and Sustainable Economic Development, ENEA Casaccia Research Center, Rome, Italy. - Embrapa Café/Lab. Biotecnologia, Área de Melhoramento Genético, Londrina, Brazil.
- VIB Nucleomics Core, Leuven, Belgium.
- These authors contributed equally: Aditi Rambani, Zhe Yu, Romain Guyot, Susan Strickler.


[제목] 이질사배체 코페아 아라비카의 게놈 및 집단 유전체학이 현대 커피 재배종들의 다양화 역사를 밝힌다.
- Coffea eugenioides와 Coffea canephora의 이질사베체 (allotetraploid) 하이브리드인 Coffea arabica는
세계 커피 생산물의 약 60%의 원천이며, 그 재배되는 액세션들은
몇차례의 개체군 병목현상(population bottlenecks)을 겪었다. - 우리는 이-반수체(di-haploid) C. arabica 액세션과
그것의 이배체 조상들인 C. eugenioides와 C. canephora의 현대적 대표들의 염색체-수준 어셈블리들을 제시한다. - 3가지 종들은 이배체(diploid) 부모들과 자손의 서브게놈들 간의 잘 보존된 게놈 구조들을 보여주며,
명확한 글로벌 서브게놈 우성 (global subgenome dominance)은 없었다. - 우리는 35만년~61만년 전의 근본적인 배수성 이벤트(polyploidy event)와,
그 후의 좁은 유전적 변이를 초래한 재배화 이전의 몇차례의 병목현상들에 대한 증거를 찾는다. - 야생 액세션들과 재배품종 조상들 간의 분리는 ~3만5백 년 전에 발생했고,
그 두 개체군들 간의 전이(migration)의 기간이 있었다. - C. canephora로 역사적으로 유전자 이입된 계열들을 포함하여, 현재의 품종들에 관한 분석은
그들의 육종 역사와, 병원체 저항성에 공헌할 수도 있는 유전자좌들을 강조하며,
C. arabica의 미래 게놈학-기반적 육종을 위한 토대를 제공한다.
배수성 (Polyploidy)는 많은 진핵생물(eukaryotic) 계통에 걸쳐 게놈 진화를 형성한 강력한 진화적 힘으로, 아마도 세계적인 변화의 시기에 적응적 이점을 제공할 수 있을 것이다 [1,2].
이러한 전체 게놈 복제 (whole-genome duplications, WGD)는 특히 식물들의 특징이며 [3], 그리고 작물 종들의 상당 부분이 배수체 (polyploid)이다 [4-11].
WGD 이후 게놈 진화에 대한 우리의 이해는 아직 불완전하지만, 그 결과에는
📌 암호화된 전이 요소 (cryptic transposable elements, TEs)의 활성화,
📌 서브게놈으로 분할된 유전자 조절 또는 분별 (gene regulation or fractionation),
📌 동조적 교환(homoeologous exchange, HE),
📌 감수분열 불안정성 (meiotic instability),
📌 심지어 핵형 변이 (karyotype variation) 등의 측면에서 게놈 충격(genomic shock)이 포함될 수 있다. [8,12-16].
아니면, 이 현상들 중 실현될 수 있는 것은 소수이거나 아무것도 없으며, 두 서브게놈들이 조화롭게 공존하여 점차적으로 새로운 배수성 수준에 적응할 수 있다 [17].
어떻든, 배수체(polyploids)의 가장 일반적인 운명은
📌 分別 (fractionation) 그리고
📌 이배체 상태로의 궁극적인 복귀(eventual reversion to the diploid state)인 것으로 보인다 [18].
연간 생산량이 천만 톤으로 추산되는 커피는 세계에서 가장 많이 거래되는 상품 중 하나이다. 가장 널리 인정받는 커피는 異質四倍體 種 (allotetraploid species)인 Coffea arabica에서 생산되며, 특히 Bourbon 또는 Typica 계통에 속하는 재배품종들과 이들의 하이브리드들에서 생산된다 [19].
🌲 C. arabica (2n = 4x = 44 chromosomes)는
각각 2n = 2x = 22 염색체를 가진 오늘날
🌲 Coffea canephora (Robusta coffee, subgenome CC (subCC))와
🌲 Coffea eugenioides (subgenome EE (subEE))의 조상들 사이의
➡ 자연 교잡 사건(natural hybridization event)으로 인해 발생했다.
근본적인 WGD는 이전에 10,000년에서 100만년 전으로 거슬러 올라가며 [20-23],
C. arabica의 로부스타-유래 하위 게놈은 북부 우간다의 C. canephora 액세션들과 가장 가깝게 관련되어 있다 [24].
아라비카 재배는 15~16세기 예멘에서 시작되었다 (Extended Data Fig. 1).
1600년경, 소위 7개의 종자가 예멘으로부터 밀수입되어 [25], 인도 C. arabica 재배품종 계통이 확립되었다.
100년 후, 네덜란드인들이 동남아시아에서 아라비카를 재배하기 시작했으며, 이로써 현대의 Typica 그룹의 설립자가 탄생했다.
1706년 암스테르담으로 선적된 나무 한 그루가 1723년 카리브해 지역에서 아라비카 재배를 확립하는 데 사용되었다.
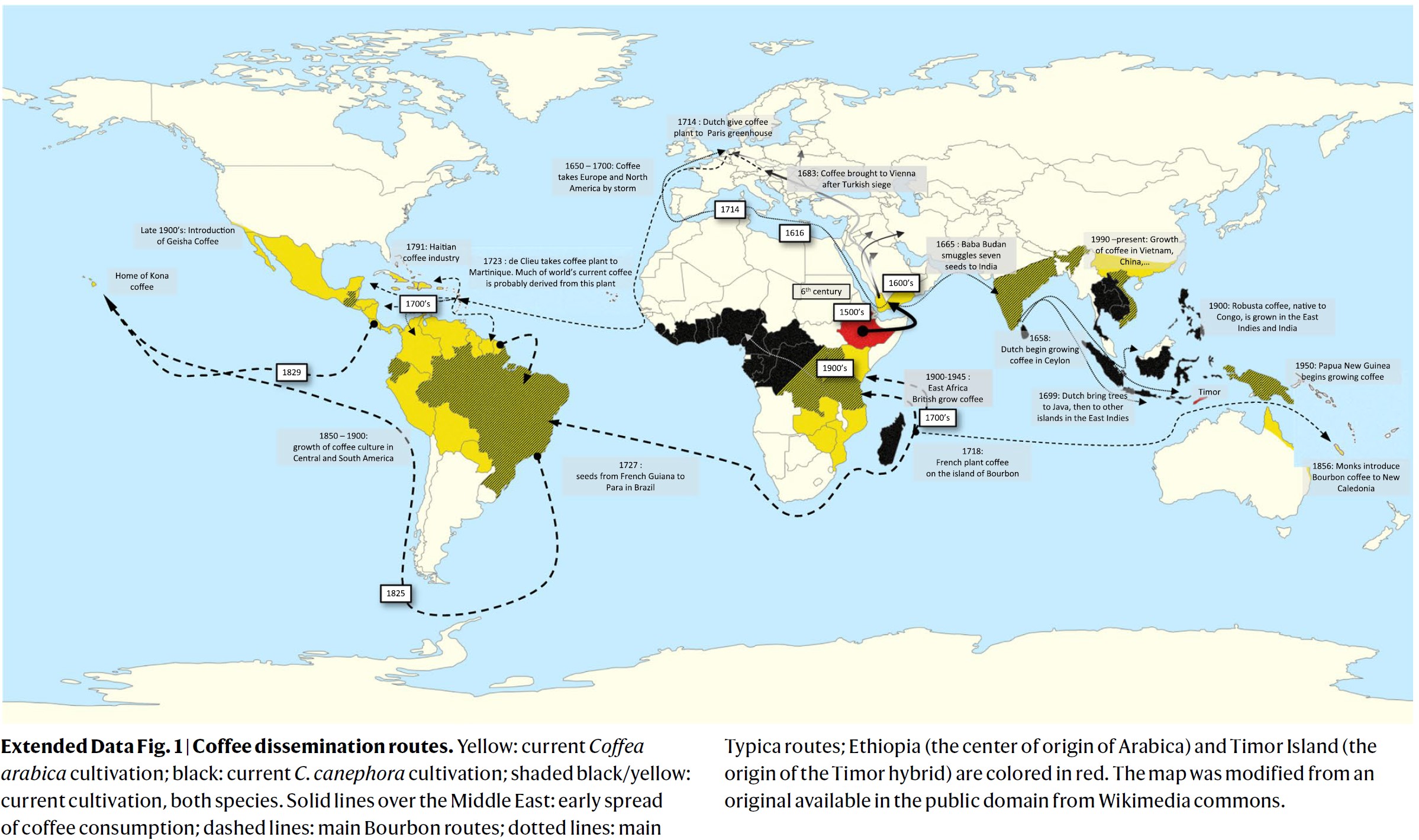
독립적으로 프랑스인들은 부르봉(Bourbon) 섬(현재의 Réunion 섬)에서 아라비카를 재배했으며 [26],
1720년까지 살아남은 나무 한 그루의 후손이 현대의 Bourbon group을 형성했다.
현대의 아라비카 재배품종들은 에티오피아의 자연림들에서 기원한 몇몇 야생 생태형(ecotypes)을 제외하고는, 티피카(Typica) 또는 버번(Bourbon) 계통의 후손이다.
그것의 最近 異質四倍體 起源 (recent allotetraploid origin)과 그 역사 동안의 강한 병목 현상(strong bottlenecks)으로 인해, 재배되는 C. arabica는 특히 낮은 유전적 다양성 (low genetic diversity)을 갖고 있으며 [20], 커피 녹병 (Hemileia vastatrix)과 같은 많은 식물 해충 및 질병에 취약하다.
결과적으로, 고전적인 Bourbon-Typica 계통은 전 세계적으로 소수의 지역에서만 성공적으로 재배될 수 있다.
다행스럽게, 1927년 티모르 섬(island of Timor)에서
H. vastatrix에 저항성을 갖는 자연발생적인
C. canephora × C. arabica 하이브리드가 식별되었다.
많은 현대의 아라비카들에는 이 하이브리드로부터 파생된 C. canephora 유전자 이입(introgressions)이 포함되어 있어, 녹 저항성을 보장하지만, 음료 품질 저하와 같은 원치 않는 부작용도 있다 [28].
현대적인 게놈 툴과 현대 품종들의 기원 및 육종 역사에 대한 상세한 이해는 기후 변화와 농업 관행에 더 잘 적응할 수 있는 새로운 아라비카 재배품종들을 개발하는데 필수적이다 [29-31].
여기에서는
♣ C. arabica와
♣ 그 대표적 조상 종인
C. canephora (Robusta) 및
C. eugenioides (이하 Eugenioides)의
➡ 염색체-수준 어셈블리(chromosome-level assemblies) 제시한다.
41개의 야생 및 재배 액세션들에 대한 전체 게놈 재배열 데이터 (Whole-genome resequencing data)를 통해 아라비카의 역사와 전파 경로에 관한 심층 분석은 물론 병원체 저항성과 관련된 후보 게놈 영역의 식별이 가능해졌다.
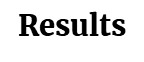
레퍼런스 개체들로서, 우리는
⊙ 이질이배체 (두 게놈성 半数体, di-haploid) Arabica line ET-39 [ref. 32],
⊙ 이전에 시퀀싱된 배가된 반수체 (previously sequenced doubled haploid) Robusta [33], 그리고
⊙ Eugenioides accession Bu-A를 각각 선정하였다.
각각 672 megabases (Mb) (Robusta), 645 Mb (Eugenioides) 그리고 1,088 Mb (Arabica)에 이르는
장편 및 단편 서열기반의 하이브리드 어셈블리들(Long- and short-read-based hybrid assemblies)이 얻어졌다.
(Methods and Supplementary Sections 2.1 and 2.2),
Hi-C scaffolding 시에, Robusta와 Arabica 어셈블리들은
11개와 22개의 僞染色體 (pseudochromosomes)로 구성되었으며,
각각 예상 게놈 크기의 82.7%와 62.5%에 걸쳐 있었다 (Table 1).
아라비카 어셈블리를 개선하기 위해 우리는
Pacific Biosciences (PacBio) HiFi 기술과 이어서
Hi-C scaffolding 을 사용하여
➡ 두 번째 어셈블리를 생성했다. (Methods and Supplementary Sections 2.2 and 2.3)
이 어셈블리의 길이는 1,198Mb였으며,
그 중 1,192 Mb (세포학적 증거에 기초하여 예측된 게놈 크기 [34]의 93.1%)가
위염색체(pseudochromosomes)에 고정되었다 (Table 1).
BUSCO (Benchmarking Universal Single-Copy Orthologs)[35]를 사용하여 평가한 유전자 공간 완전성 (Gene space completeness)은 모든 어셈블리에서 >96%였다. 중요한 것은 BUSCO 유전자의 93.2%가 HiFi 어셈블리에서 복제되었으며 (Table 1), 이는 異質倍數性 이벤트(allopolyploidy event)에서 발생한 유전자 복제 (gene duplicates)의 대부분이 유지되었음을 가리킨다.
Robusta 및 Eugenioides 게놈은 각각 67.5% 및 59.7% TEs를 포함하고 있으며 (Supplementary Section 3.2),
Gypsy long terminal repeat (LTR) retrotransposons이 두 종 간의 차이의 대부분을 차지한다.
이 차이는 두 개의 아라비카 서브 게놈(각각 Robusta 및 Eugenioides 조상에서 유래한 subCC 및 subEE)에서 크게 감소(63.1% 및 63.8%)했으며, 이는 HE를 통한 TE transfer를 가리키는 것일 수 있다.
Robusta는 Eugenioides보다 훨씬 더 최근의 LTR TE insertion elements를 포함하고 있다.
다시 말하지만, 두 아라비카 서브 게놈들은 두 조상 게놈들(progenitor genomes) 보다 최근 LTR TE insertions에서 서로 더 큰 유사성을 나타냈다.
LTR TE mobilization에 이은 아라비카 이질배수체화 (allopolyploidization)에 대해 주요 증거는 발견되지 않았지만, 대조적으로 담배의 경우에서는 관찰된 바 있지만 [36], 브래시카(Brassica, 배추속 식물) 합성 이질사배체들과는 유사하다 [37].
대신에, 관찰된 아라비카 게놈 진화는 애기장대(Arabidopsis) 하이브리드들에서 보이는 ‘harmonious coexistence’ pattern (조화로운 공존패턴) [38]을 더 가까이 따른다 [17,39].
고품질 유전자 주석 (High-quality gene annotations)에 이어 특정 유전자군을 수작업으로 큐레이션 (manual curation)한 결과 (Supplementary Sections 3.1–3.4), Robusta, Eugenioides, PacBio Arabica 및 Arabica HiFi 어셈블리에 대한 각각 28,857, 32,192, 56,670 및 69,314개의 유전자 모델이 생성되었다 (Table 1).
전체적으로, 로부스타의 ~97%와 아라비카 HiFi 유전자 모델의 99.6%가 僞染色體들에 배치되었으며, 각각 33,618개와 35,449개는 서브게놈 subCC 및 subEE에 해당된다 (Table 1).
BUSCO로부터의 주석 완성도(Annotation completeness)는 Eugenioides와 Robusta의 경우 95% 이상이었고, Arabica HiFi의 경우 97.3%에 달했다.
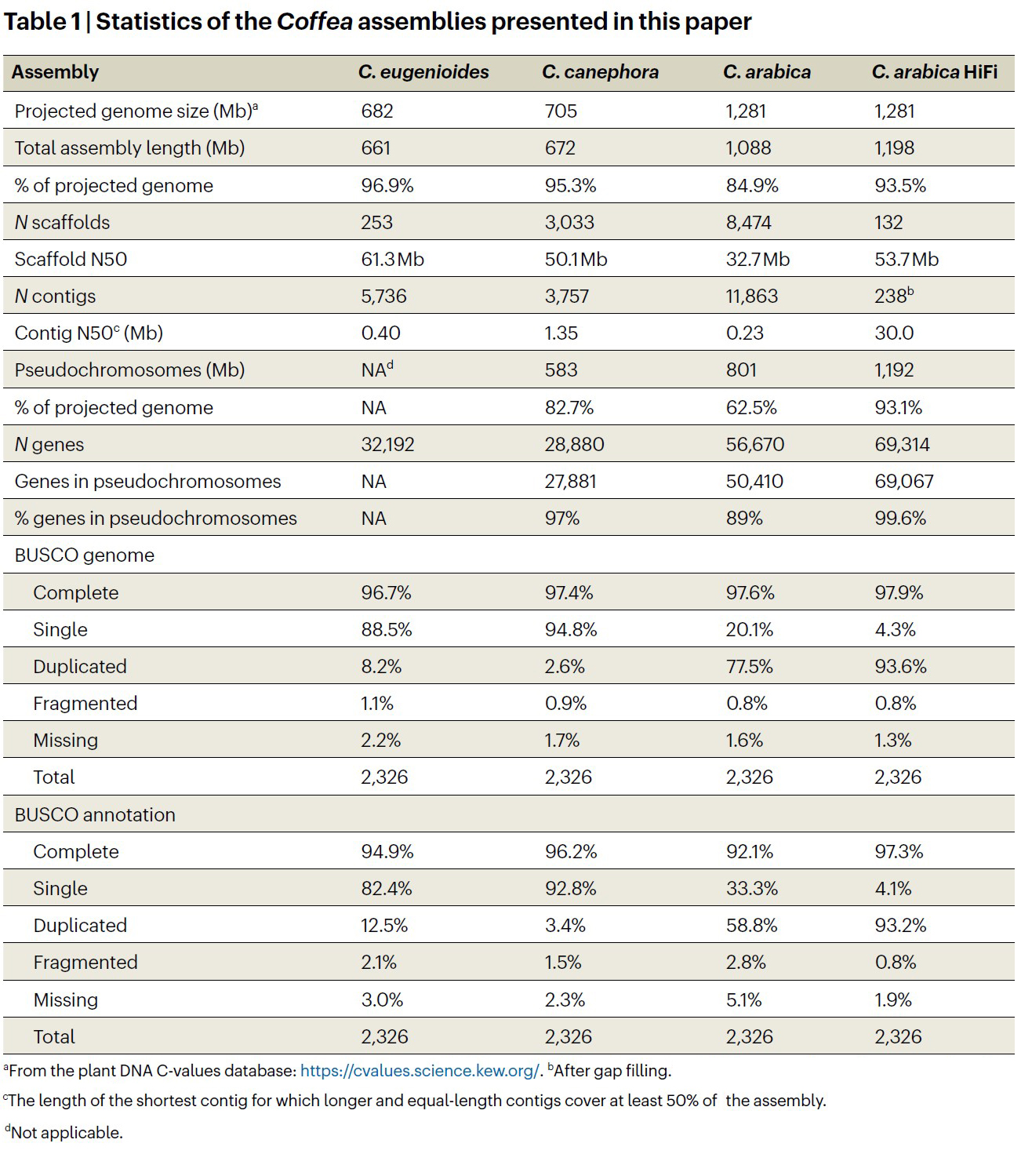
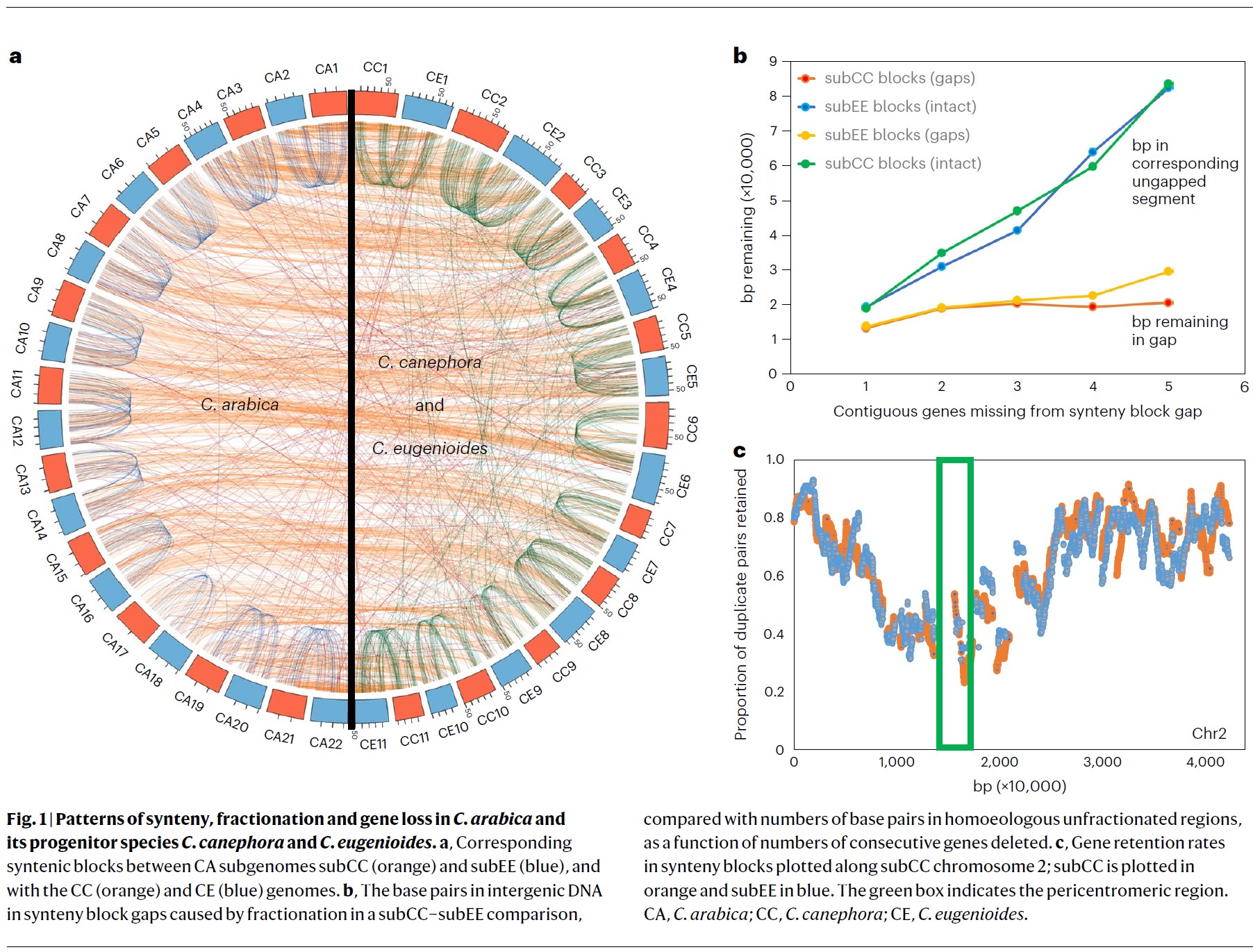
Fig. 1 : C. arabica 그리고 그 조상종 C. canephora 및 C. eugenioides에서의
신테니, 분별 그리고 유전자 결실의 패턴
a. CA 서브게놈들 subCC (오렌지색) 및 subEE (청색), 그리고 CC 게놈 (오렌지색)과 CE 게놈 (청색) 간의
상응하는 신테닉 블록들.
b. 결실된 연속적 유전자 수의 함수로서, subCC–subEE 비교에서 분별에 의해 유발된
신테니 블록 갭들에서의 유전자간 DNA에서의 염기쌍들,
동조적인 분별되지 않은 영역들에서의 염기쌍들의 수와 비교.
c. subCC 염색체 2를 따라 플롯된 신테니 블록들에서의 유전자 보유율;
subCC는 오렌지색으로 그려져 있음. 녹색 박스는 중심절 주위 영역을 가리킴.
CA ➡ C. arabica; CC ➡ C. canephora; CE ➡ C. eugenioides.
🚀 Synteny
➡ 복수의 유전자가 물리적으로 동일 염색체 또는 동일 게놈 영역 위에 존재하는 상태
➡ 두 종에서 여전히 보전되고 있는 부위
➡ Synteny란 어떤 주어진 종의 동일한 염색체에서의 2개 이상 유전자들의 존재를 정의한다.
Synteny 상태는 어떤 염색체 축을 따른 유전자들의 상대적 순서에 관한 정보나
한 연색체 상의 유전자들 간의 거리에 관한 정보를 필요로 하지 않는다.
➡ Dense gene-based marker maps와 시퀀스된 게놈들의 이용가능성으로 인해,
synteny라는 용어는 두개의 연관된 종들의 조상-관련적 연결 그룹들에서,
동일한 syntenic genes의 존재를 가리키며, 따라서 conserved synteny의 상태를 설명하며,
비교 게놈학 맥락에서 지배적으로 사용되고 있다.
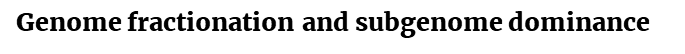
아라비카 subCC 및 subEE를 Robusta 및 Eugenioides의 것들과 비교한 결과,
⊙ 염색체 수,
⊙ 동원체 위치 (centromere position) 및
⊙ 염색체당 유전자 수 측면에서
➡ 높은 보존성 (high conservation)이 나타났다 (Fig. 1 and Supplementary Section 4).
감마 古육배체 현상(gamma paleohexaploidy event)에 따른 유전자 손실 패턴은, 그들의 초기 종들이 분리 (initial species split)된 이후 4~6백만 년 동안 Robusta와 Eugenioides 사이에 높은 구조적 보존 (high structural conservation)을 나타냈다 [22,23] (Supplementary Section 4).
마찬가지로, 두 개의 아라비카 서브 게놈들의 구조는 서로간에 고도로 보존되었으며, 아라비카의 근본적인 이질사배체성 사건 (allotetraploidy event) 이후, BUSCO 유전자 중 ~5%만이 이배체 상태로 되돌아갔다 (Fig. 1a and Table 1).
신테닉 비교(Syntenic comparisons, 동염색체 비교)를 통해,
두 하위 게놈에 걸쳐 비슷한 비율로 한 번에 하나 또는 여러 개의 유전자를 제거하는
게놈 절제 사건(genomic excision events)이
➡ 배수성 사건(polyploidy event) 전후에
게놈 단편화(genome fragmentation, 게놈 斷裂, 分別)의 주요 원동력이었음이 밝혀졌다
(Fig. 1b and Supplementary Section 4).
分別 (斷裂. Fractionation)은 ➡ 주로 동원체 주위 영역(pericentromeric regions)에서 발생하는 반면,
염색체 암(chromosome arms)은 ➡ 더 적당한 유사유전자 결실 (paralogous gene deletion)를 나타냈다
(Fig. 1c and Supplementary Section 4).
아라비카 異質倍數性 事件(allopolyploidy event)은
➡ 게놈 분별 속도(rate of genome fractionation)에 영향을 미치지 않은 것으로 보이며,
➡ 그 이벤트 후 조상 종과 아라비카 서브 게놈의 삭제(deletions)를 비교할 때, 게놈 단열 속도는 거의 일정하게 유지되었다.
투여량-균형 가설(the dosage-balance hypothesis)을 서포트하기 위해 [40], 높은 복제 보유율 (high duplicate retention rates)을 가진 서브 게놈 영역들은 아라비카 WGD로부터 유래한 유전자들의 경우에 상당히 풍부했다 (Fisher exact test, P < 2.2 × 10-16).
대조적으로, 낮은 복제 보유율 영역(low duplicate retention rate regions)은 소규모 (직렬 tandem) 복제(duplications)으로부터 유래하는 유전자와 상당히 겹쳤다 (Supplementary Table 1).
높은 보유율(high retention rates)의 유전자는
➡ 'cellular component organization or biogenesis (세포 구성요소 조직 또는 생물 발생)’,
➡ 'primary metabolic process (1차 대사 과정)',
➡ 'developmental process (발달 과정)’ 및
➡ 'regulation of cellular process (세포 조절 과정)'과 같은 유전자 존재론(Gene Ontology, GO) 카테고리에서
풍부했으며,
낮은 보유율 유전자(low retention rate genes)는
➡ 'RNA-dependent DNA biosynthetic process (RNA-의존적 DNA 생합성 과정)' 및
➡ 'defense response(방어 반응)’ (두 서브게놈 모두에서),
➡ 'spermidine hydroxycinnamate conjugate biosynthetic process
(스페르미딘 하이드록시신나메이트 접합체 생합성 과정)’ (식물 방어에 관여[41]) 및
➡ 'plant-type hypersensitive response (식물형 과민 반응)'(subEE에서)와 같은
카테고리들에서 풍부했다 (Supplementary Tables 2–5).
서브 게놈들 간의 가능한 발현 편향(expression biases)을 연구하기 위해,
우리는 합성 유전자 쌍들(syntelogous gene pairs)을 식별하고
아라비카 서브 게놈에서 HEs를 나타내는 쌍을 제거했다
(아래 ' Origin and domestication of Arabica coffee' 참조) [42] (Supplementary Section 5).
전반적으로, 유의미한 전체 서브 게놈 발현 우세(global subgenome expression dominance)는 관찰되지 않았다
(Supplementary Tables 6 and 7).
그러나 유전자군(gene families)은 N-methyltransferase (NMT), terpene synthase (TPS) and fatty acid desaturase 2 (FAD2) 계열들과 같이 컵 품질에 기여하는 여러 인코딩 효소(encoding enzymes)를 포함하여 정기적으로 모자이크 패턴의 발현(mosaic patterns of expression)을 나타냈으며, 모두 최근 연구에서와 같이, 두 서브 게놈들(Extended Data Fig. 2) 중 하나에서 더 많이 발현되는 어떤 유전자들을 가지고 있다 [43].
유사한 유전자군별 패턴들(gene family-wise patterns)은 유채씨(rapeseed)[10] 및 목화(cotton) [44]와 같은 진화적으로 최근의 다른 배수체(polyploids)에서도 발생하며, 이 배수체들도 이배체 상태로 다시 전환하는 초기 단계에 있다.

Extended Data Fig.2 콩 품질 특성들에 기여하는 예시적 아라비카 유전자 패밀리들의 조성과 발현
a. 카페인 (좌), 테르펜 (중), 그리고 불포화지방산 (우)의 생합성도.
b. 카페인 생합성 (좌), 테르펜 합성 (TPS) (중), 그리고 지방산 탈수소효소 2 (FAD2) (우)를 조절하는
N-methyltransferases (NMTs)에 대한 CA 유전자들의 계통발생 및 과실 발달 중의 발현.
K7 재배품종의 3가지 다른 과실 성숙단계들 (녹색, 황색, 적색)로부터의 3가지 생물학적 복제물들에 대해
RNA 시퀀싱이 수행되었다.
c. 게놈 차원의 NMT (좌), TPS (중), FAD2 (우) 유전자 트리들과 과실 발달 중의 발현 패턴들,
두 서브게놈들에 위치한 유전자들은 포트 컬러로 표시된다; subCC (red) and subEE (blue).
Arabidopsis 유전자들은 갈색이다.
원형 트리들에서 회색 쐐기들은 b에 나온 트리 부분들을 강조한다.
XMT: xanthosine methyltransferase;
MXMT: 7-methylxanthine methyltransferase;
DXMT: 1,7-dimethylxanthine methyltransferase;
MTL: N-methyltransferase-like;
FS: (E,E)-a-farnesene synthase;
GS: Geraniol synthase;
IS: Isoprene synthase;
MS: myrcene synthase;
TS: (-)-a-terpineol synthase;
FAD2: Fatty acid desaturase 2.
유전자 발현은 컬러 스케일로 표시되며, 노란색 (positive)부터 red를 거쳐 blue (negative)까지,
log10 transcripts per million (log10 TPM)의 단위로 표시.
회색부분들은 Arabidopsis 유전자들의 발현 데이터 부재를 가리킨다.

Fig. 2 | Population history of C. arabica.
a. 리시퀀스된 야생 C. arabica 액세션들의 지리적 기원 (적색 플레이스홀더들).
액세션 이름들이 c.에 나와 있다.
빨간색 화살표는 역사적 시대에 예멘으로의 개연성 있는 이주 경로를 가리킨다.
b. subCC (left) 및 subEE (right)의 C. arabica 액세션들의 조상 개체군 배정.
개체들 사이의 관계들이 독립적인 SNPs로부터 얻어진 계통발생 트리들로 그려져 있다.
트리의 확대도들은 Supplementary Fig. 37을 참조.
c. A의 좌하단부를 확대한 것,
subCC (top) and subEE (bottom)에서의 각 액세션들의 admixture 값들을 보여준다;
색상들은 b에서의 분석과 상응한다.
d. SMC++를 사용하여 추론된 야생 액세션 및 재배 액세션들의 개체군 사이즈들이 ~350 and 1ka에서의
유전적 병목들을 제시한다 (non-admixed 야생 개체들로 제한됨).
e. FastSimcoal2 아웃풋이며, ~30.5 ka 개체군 분리에 이어 ~8.9 ka까지 개체군들 간 이주기간을 제시한다.
이 시기는 SMC++로 계산된 비슷한 시대에 재배종들에서의 증가된 개체군 다양성과 상응한다.
타임라인의 녹색 사각형은 ‘windows of opportunity’을 보여주는데, 아라비아 반도로의 인간의 이주가
발생했을 수 있는 아프리카 대륙에 예멘이 연결되었던 시대이다.
퍼플색 사각형은 지난 빙하기를 보여준다. M, migration; OAE, out-of-Africa event.
f. Orientagraph를 사용한 방향성 유전자 흐름 분석은 두 가지 가설들을 제시한다:
모든 재배종들의 공유 조상 개체군으로부터 에티오피아 야생 개체들 (subCC)로 유전자의 흐름, 또는
티피카 혈통의 에디오피아(subEE)로의 유전자 흐름.
a와 c의 지도들은 각각 Google Earth와 Google Maps으로 만들어졌다.

아라비카의 진화 역사에 대한 게놈적 관점을 얻기 위해, 우리는
♣ 3개의 Robusta,
♣ 2개의 Eugenioides, 그리고
♣ 41개의 Arabica를 포함하여 46개 액세션들의 서열을 분석했다.
아라비카 액세션들에는
♣ 런던 Linnaean Society에서 친절하게 제공한 18세기 타입 견본,
♣ 다양한 육종 이력을 지닌 12개 재배품종들,
♣ Timor hybrid 그리고 그것의 아라비카와의 역교배(backcrosses) 5개,
♣ the Great Rift Valley의 동쪽과 서쪽으로부터 수집된 17개의 야생 액세션들과
♣ 3개의 wild/cultivated accessions가 포함되어 있다 [45,46] (Supplementary Table 8 and Fig. 2a).
서브 게놈 사이의 HE는 최근 여러 배수체(polyploids)에서 관찰되었다 [8,10,42].
아라비카는 일반적으로
📌 상동 염색체(homologous chromosomes)의 2가 쌍(bivalent pairing)과
📌 二染色體 유전(disomic inheritance)을 나타내지만 [47],
📌 그 서브 게놈들이 높은 유사성을 공유하기 때문에,
➡ 간헐적으로 同祖 페어링 (homoeologous pairing) 및 교환이 발생할 수도 있다.
따라서 우리는 아라비카 액세션들에서의 HE의 정도와 게놈 진화에 대한 기여 가능성을 조사했다.
전반적으로 모든 액세션들은 엽록체 관련 기능이 풍부한 유전자를 포함하는 염색체 7의 한쪽 끝에서 subEE에 대한 고정 대립유전자 편향(fixed allele bias)을 공유했다 (Extended Data Fig. 3a, Supplementary Section 5 and Supplementary Table 9).
아라비카 色素體 게놈(plastid genome)은 Eugenioides에서 유래하므로 [48], 엽록체에 위치하는 단백질을 인코딩하는 핵과 엽록체 유전자 사이의 호환성 문제로 인해 이 영역의 HE가 선택되었을 가능성이 높다 [49].
놀랍게도, 하나의 액세션 (BMJM)을 제외한 모든 액세션들이 유의미한(Bonferroni-adjusted P values < 0.0005 ; chi-squared test, each d.f. = 1) subCC에 대한 3:1 대립유전자 편향(allelic biases)을 나타냈다.
야생 아라비카와 재배 아라비카 모두에 존재하는 고도로 일치하는(concordant) HE 패턴(Extended Data Fig. 4)은
(1) 대립유전자 편향이 육종과 관련이 없는 적응 특성이며,
(2) 아마도 근본적인 이질배수체 사건(the founding allopolyploidy event) 직후에,
표본 추출된 모든 액세션들의 공통 조상에서 유래했음을 시사한다.
소수의 액세션들만이 공유하는 일부 교환들은 아마도 더 최근에 시작되었을 것이다 (Extended Data Fig. 3b).
더 최근의 HE 사건들이 일부 재배품종들에서도 발견되었으며, 염색체 1에서의 단일한 큰 교차(single large crossover)로 인해 subEE에 대한 편향을 보인 BMJM을 제외하고 subCC에 대한 편향도 나타났다 (Extended Data Fig. 3a).
미래 조사를 위한 흥미로운 한 가지 가설은 아라비카와 같이 다양성이 낮은 배수체 종(polyploid species)에서, HE가 밀접하게 관련된 액세션들 중에서 관찰되는 표현형 변이에 대한 주요 기여자가 될 수 있다는 것이다 [50].

다음으로 우리는 각 서브 게놈에 대한 개체군 유전 통계를 연구했다 (Supplementary Table 10).
17개의 야생 샘플들은 낮은 유효 개체군 크기를 나타내는 낮은 게놈 다양성(low genomic diversities)을 보인 반면,
음의 Tajim’s D는 아마도 하나 이상의 개체군 병목 현상(population bottlenecks)에 이어지는 확장하는 개체군 (expanding population)을 제시했다.
낮은 고정 지수(low fixation index, FST) 값에서 알 수 있듯이, 재배품종들과 야생 개체군 샘플들은 유사한 유전적 다양성을 가졌다.
재배품종들에서는, 뉴클레오티드 다양성(nucleotide diversities)은 야생 개체군들보다 약간 낮았으며, Tajima’s D 스코어는 덜 부정적(less negative)이어서, 재배화(domestication) 중에 사소한 병목 현상과 그에 따른 개체군 확장 만 발생했음을 나타낸다.
SNP 트리 추정 및 ADMIXTURE 분석 (Fig. 2b)은 subCC의 경우 3개 개체군 솔루션을 식별했다:
♣ Typica-Bourbon cultivars (Population 1),
♣ wild accessions (Population 2) 그리고
♣ Timor hybrid-derived cultivars (Population 3).
오래된 BMJM과 최근 확립된 Geisha 재배품종들은 두 서브 게놈 모두에서 혼합 상태(admixed states)를 보여주었는데, 이는 야생 액세션들의 약 절반과 비슷하다.
인도 재배품종들은, 이전 연구와 일치하여, Typica와 Bourbon 변종을 모두 포함했다 [20].
Linnaean 샘플은 재배품종들과 함께 그룹화되어, the Dutch East Indies에서 기원했다는 가설을 뒷받침한다 [25].
보완적인 주성분 분석(principal component analysis, PCA) (Extended Data Fig. 5)은 ADMIXTURE analysis와 일치했다.

야생 액세션들에서, 두 하위 게놈 모두 SMC++(ref. 51) 모델링에서 2개의 population bottlenecks (Fig. 2d)을 동시에 보여주었다.
세대 시간을 21년으로 가정하면 [52], 가장 오래된 bottleneck은 약 35만년 전(ka)에 갑자기 시작되어 아프리카 습윤 기간이 시작되는 약 15ka에 끝났으며, 이때 기후 조건은 아라비카 성장에 더 유리했다.
보다 최근의 bottleneck은 약 5,000년경에 점차적으로 시작되어 오늘날까지 지속된다. 그러나 재배되는 액세션들은, 가장 오래되었지만 가장 최근에는 bottleneck은 나타내지 않았다.
부분적으로 이러한 차이로 인해, 우리는 FastSimcoal2 (ref. 54)를 사용하여, 아라비카 개체군의 역사를 모델링하여 야생 개체군과 재배품종들을 두 개의 별도 계통으로 모델링했다.
베스트-피팅 모델 (Fig. 2e)에서, 야생 개체군은 1,450세대 전(~30ka), 즉 마지막 빙하 최대치 (the last glacial maximum) 이전에 재배품종 창립 개체군(founding population)으로부터 분리될 것으로 예측되었다. 오리지날 founding event는 혼합되지 않은 야생 개체들을 사용하여 분석되었으며, 350ka에서 조상 개체군 병목 현상(ancestral population bottleneck)이 나타났다 (Extended Data Fig. 6a).
📌 유전자 분별 (gene fractionation),
📌 비동일 돌연변이들(nonsynonymous mutations)의 분포(Extended Data Fig. 6b) 그리고
📌 보정된 SNP 트리 (Fig. 2b)를
기반으로 한 분기 추정(Divergence estimates)은
➡ 이질배수체 형성 사건 (the allopolyploid founding event)이
이전 추정과 가까운 610ka에서 발생했음을 시사했다 [22,23].
한편, 350ka bottleneck은 SMC++ 분석에서 발견된 것과 일치한다 (Fig. 2d).
➡ 따라서 우리는 610-350ka를 배수체화 사건(polyploidization event)의 가능한 시간 범위로 생각한다 (Fig. 2e).
야생 및 재배-전 계통들은 ~ 8-9 ka까지 일부 유전자 흐름 (이동 측면에서)을 유지했으며, 이는 유효 개체군 사이즈의 모델화된 증가에 기여했을 수 있다 (Fig. 2d, e).
이 데이터는 현대의 재배되는 개체군의 정확한 기원을 식별할 수 없었지만(다음 섹션 참조), 야생과 재배되는 액세션들 간의 길어진 이동(migration) 기간은 이들이 아프리카의 Great Rift Valley의 양쪽과 같이, 비교적 작은 지리적 거리에 의해 분리되었음을 제시한다 (Fig. 2a–c).
또한 경작되는 계통이 예멘까지 확장되었을 수도 있고, 아프리카 습윤기가 끝날 무렵 해수면 상승으로 인해 the Bab al-Mandab strait (예멘과 아프리카를 분리하는 바브 알-만다브 해협)이 넓어지면서 [55] 두 개체군 사이의 이주가 중단되었을 수도 있다.
토착 아라비카 개체군이 예멘에 존재하며 [56], 이는 이 가설을 뒷받침할 수 있을 것이다.
Typica 및 Bourbon cultivars와 함께 Linnaean 샘플은, 이 두번째 개체군으로부터 유래하는데, 이는 SNP, ADMIXTURE 그리고 PCA 분석들에 의해 제시된 바와 같이 (Fig. 2b and Extended Data Fig. 5), 예멘에서의 경작을 확립하는 데에도 사용되었을 것이다.
결론적으로, 우리의 분석은 Coffea 개체군들에 존재하는 근친교배(inbreeding)가 합체(coalescence) 추정을 가속화할 것이라는 점을 고려할 때,
➡ 아라비카 이질배수체 사건(allopolyploidy event)이 610~350ka 사이에 발생했음을 시사한다 [57,58].
20ka (ref. 20)와 같은 보다 최근의 시기를 제안하는 초기의 연구는 재배 혈통 및 야생 혈통에서 개체군 bottlenecks의 혼동 효과 (confounding effects)로 인해 생기는 과소추정일 수도 있을 것이다.

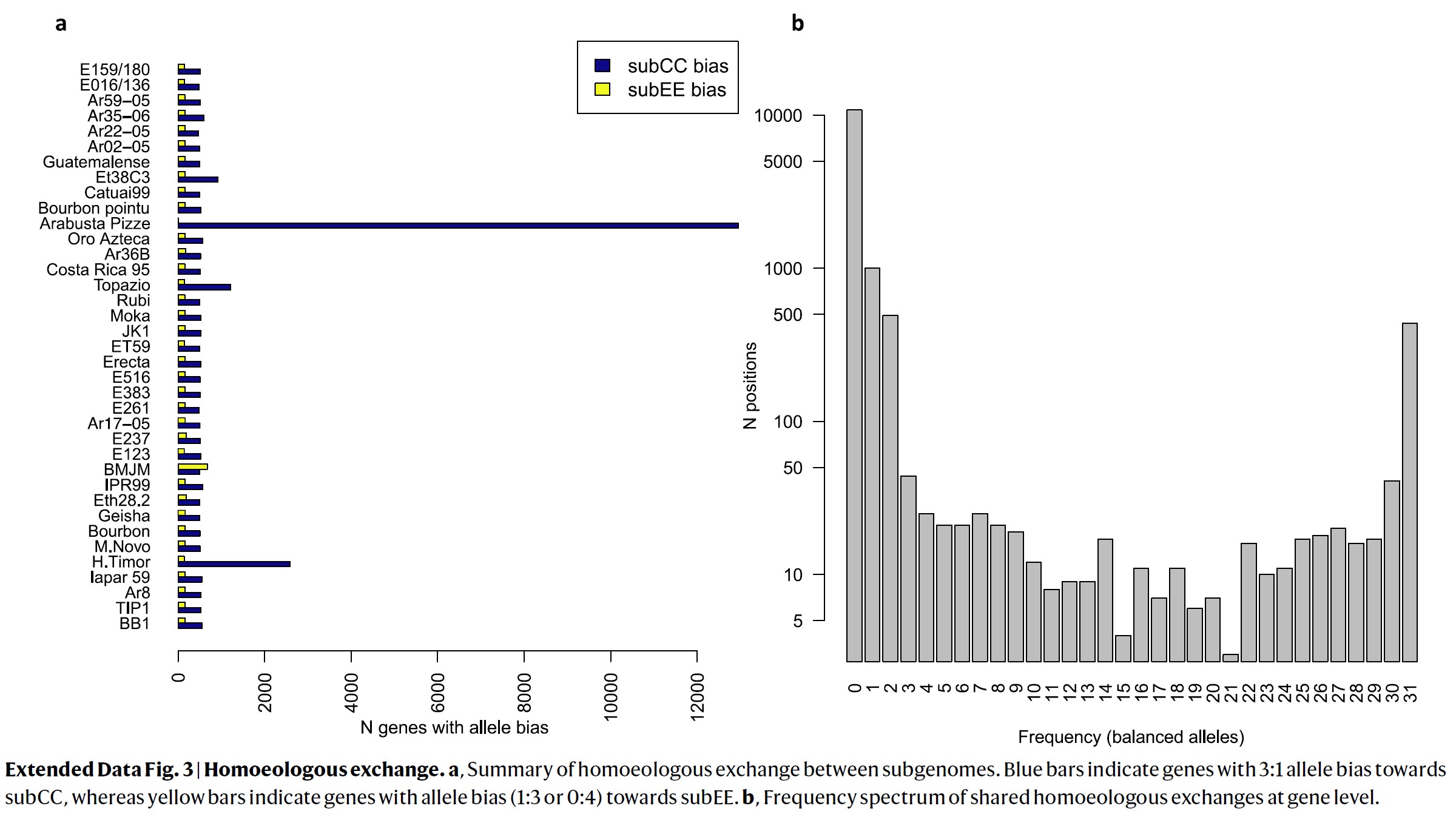
몇몇 우리의 아라비카 재배품종들의 알려진 육종 역사는
Kinship-based INference for Gwas (KING) [59] (Fig. 3)를 사용하여
아라비카 혈통(pedigree)을 추론하기 위한 최적의 스탠다드를 제공했다.
이 방법은 Bourbon과 Typica 그룹 재배품종들과 subCC에서의 Bourbon-Typica crosses 사이의 관계를
정확하게 식별했다.
대조적으로, subEE 혈통 (pedigree)은 아마도 그 해당 서브 게놈에서의 HE로 인해, 더 낮은(2차) 차수의 관계를 나타냈다 (Extended Data Fig. 7).
Timor hybrid-derived 액세션들은 subCC에서의 주요 계열 재배품종들과 유의한 관계를 나타내지 않았지만 (아마도 일배체형 블록 (haplotype blocks)을 파괴한 이 서브 게놈의 로부스타 유전자 이입으로 인해 발생했을 가능성이 있음; 아래 참조),
subEE는 Typica 및 Bourbon 그룹들 모두와 2차 관계(second-degree relationships)를 나타내 (Fig. 3 and Extended Data Fig. 7), subEE가 실질적인 이입을 받지 않았음을 확인해준다.
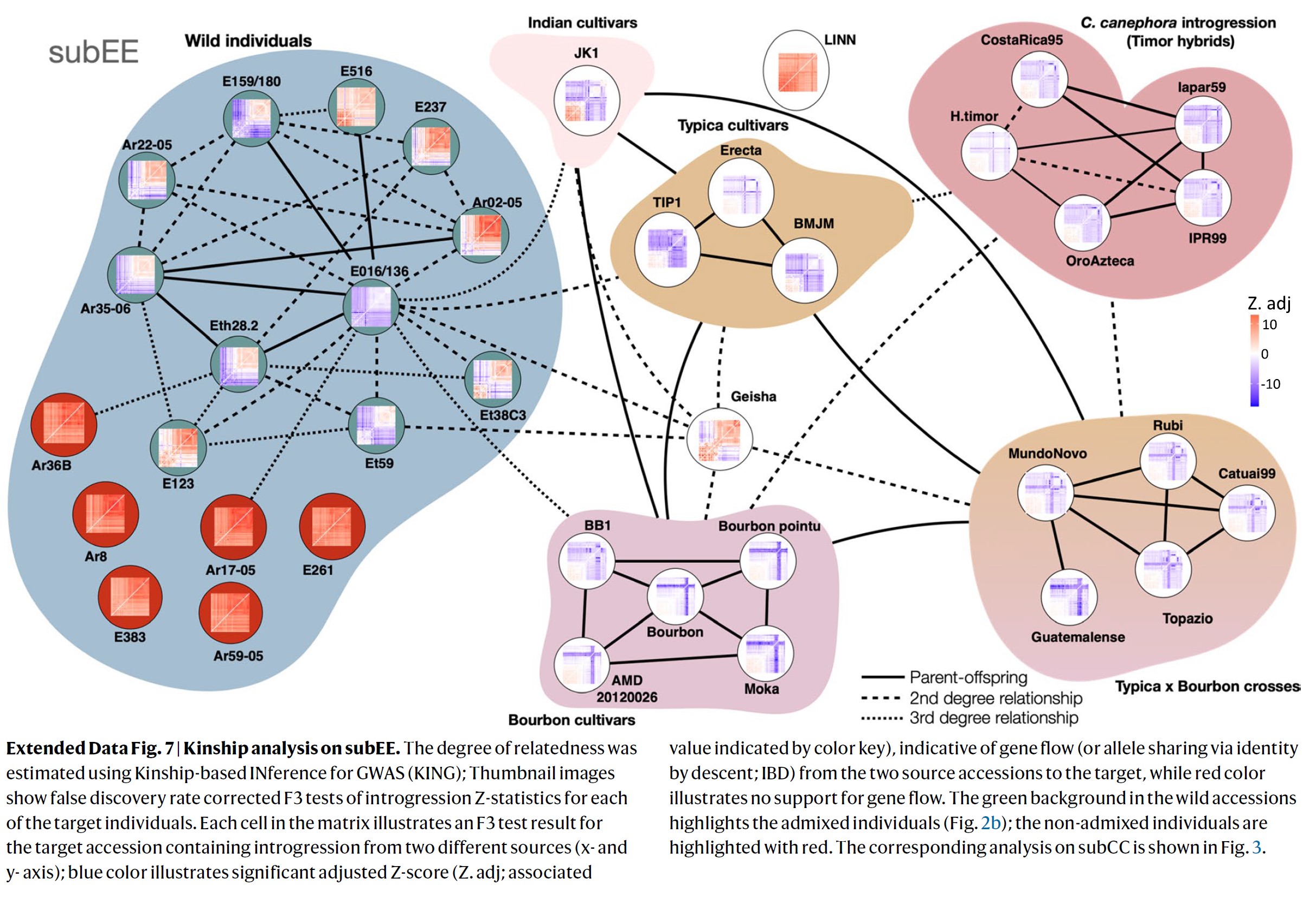
흥미롭게도 Typica, Bourbon 및 JK1 개체들도 1차 관계가 되어 있어 직접적인 부모-자손 관계를 제시한다.
그들의 공유적인 예멘 기원을 확인하는 것 외에도, 이 발견은 또한 예멘 생식질의 제한된 유전적 다양성을 강조한다.
또한, 기존 재배품종 계열들 JK1 (인도), Electa (인도네시아 Typica), BMJM (Caribbean Typica), TIP1 (Brazilian Typica) 그리고 BB1 (Brazilian Bourbon)은 밀접하게 관련된 야생 혼합 액세션들의 E016/136을 중심으로 한 클러스터와 2차 이상의 관계를 보여주었다 (Fig. 2b).
최근 확립된 게이샤 재배품종은 야생 혼합 개체들과 Bourbon 및 Typica 그룹과 유사한 관계를 보여, 공통의 기원을 시사한다.
흥미롭게도, 혼합된 야생 액세션 E016/136은 야생 개체군과 재배 개체군 모두와 밀접하게 관련되어 있었다.
지리적 기원들을 비교해 보면, the Great Rift Valley 동쪽 지역의 야생 개체들은 어느 정도 혼합되어 밀접하게 상호 연관되어 있는 반면, 서쪽 지역에서는 혼합된, 관계된 개체들이 대부분 Gesha 지역 주변에 집중되어 있었다 (Figs. 2c and 3).
재배품종들에 가장 가까운 E016/136 admixed accession은 몇 가지 야생 액세션들과 1차 관계를 보여 주었으며, 그 중 Ar35-06과 Eth28.2만이 야생 개체군의 순수한 대표자였다 (Fig. 2b).
따라서 이 두 액세션들은 우리 표본에서 재배되는 아라비카의 가상의 진정한 야생 부모와 유전적으로 가장 가깝고, E016/136은 중간 형태를 나타낸다.
Ar35-06은 현대의 Geisha 재배품종의 기원에 가까운 게샤(Gesha) 산 근처에서 수집되었다.
전체적으로 이러한 데이터는 게샤(Gesha) 지역이 재배화에 적합한 야생 액세션들의 핫 스폿임을 나타낸다.
혼합된 야생 샘플들(Admixed wild samples)은 야생으로부터 수집하기 전이나 후에 발생한 최근 교배 사건 (hybridization event)으로부터 유래했을 수 있다.
세 번째 대안은 예멘 개체군(따라서 재배품종들)이 Great Rift Valley 동쪽 또는 게샤(Gesha) 지역의 혼합 개체군으로부터 유래했다는 것이다.
Orientagraph [60] (Fig. 2f)을 사용한 혼합 패턴 분석(Analysis of admixture patterns)은
subCC에서의 Bourbon 및 Typica 계통들의, 그리고 subEE에서의 Typica 계통의 공통 조상과의 혼성화(hybridization)를 제시했다.
최근 혼성화의 경우, 도입된 일배체형들(haplotypes)은 긴 연속 블록으로 존재하는 반면 (100년 전에 발생한 티모르 혼성화에서처럼), 오래된 이벤트들의 경우 교차(crossing-over)로 인해 블록이 더 단편화(more fragmented)되었을 것이다.
the distance fraction (df) 통계량 [61]를 사용한 분석에서는, 후자가 해당되는 것으로 나타났으며 (Extended Data Fig. 8), 이는 야생 액세션들 간의 혼합 사건(admixture events)이 그리 최근에 발생하지 않았음을 나타내며, 이는 우리의 세 번째 가설을 뒷받침한다.

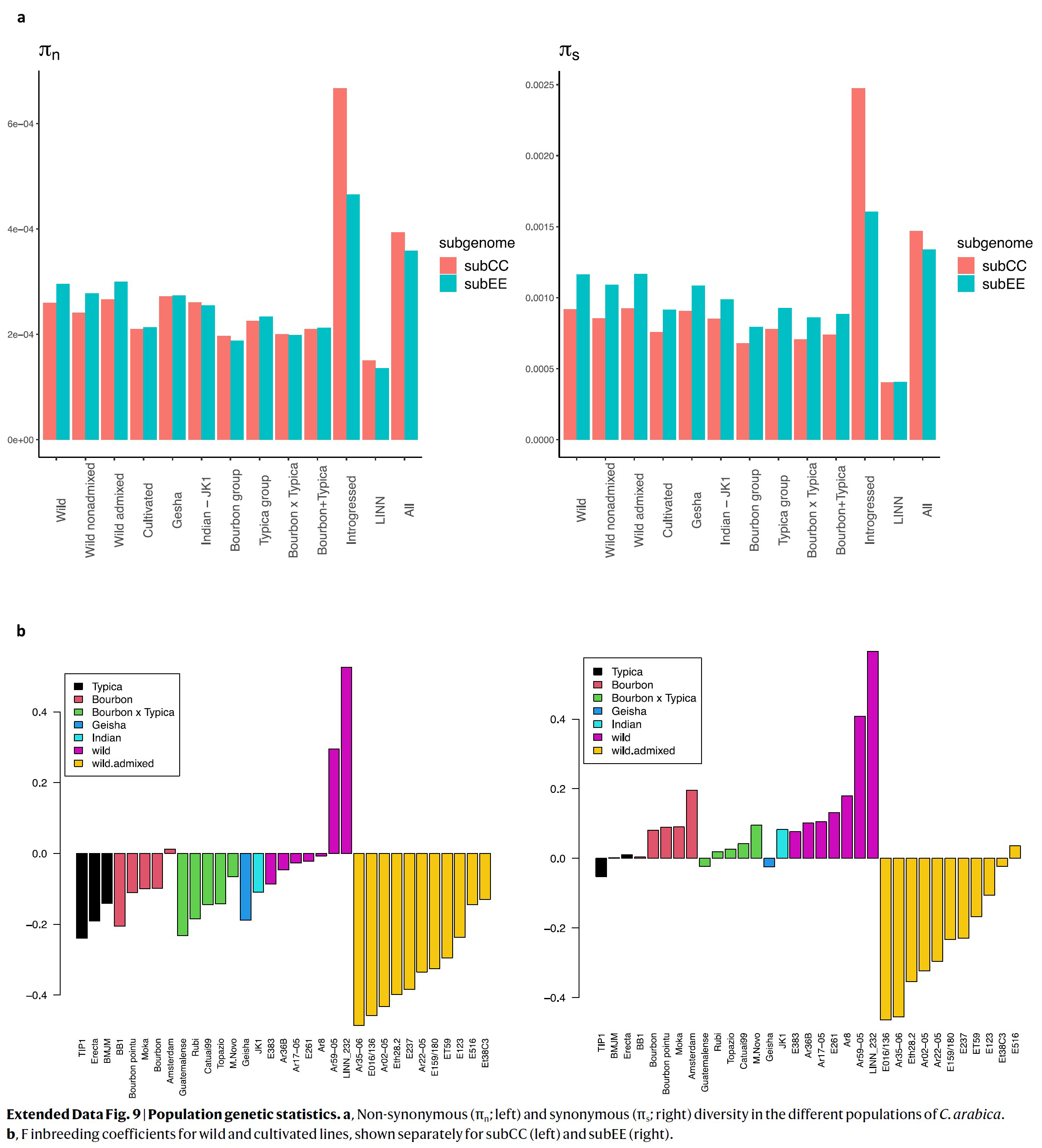
재배화(Domestication)와 경작(cultivation)은 보통 높은 야생 다양성(high wild diversity)을 기반으로 하는 강력한 개체군 bottlenecks를 수반하며, 이로 인해 재배품종들에서의 유전적 다양성이 감소한다 [62].
그러나 아라비카 뉴클레오티드 다양성(nucleotide diversity)은 야생에서 이미 매우 낮았는데, 이는 아마도 더 앞선 병목 현상(earlier bottlenecks)의 결과일 것이지만 (Fig. 2d, e), 재배되기 전의 계통(pre-cultivated lineage)에서는 약간만 감소했다 (Extended Data Fig. 9a).
Bourbon은 Typica보다 다양성이 낮았는데, 이는 아마도 이 그룹의 알려진 single-individual bottleneck 때문일 것이다.
또한, 야생 및 재배되는 액세션들에서의 근친교배 계수(inbreeding coefficients)는 비슷했으며(Extended Data Fig. 9b), 재배화된 종에 대한 일반적인 기대와는 달랐다 [62].
재배품종들에서의 정화 선택 경로(pathways under purifying selection)를 찾기 위해,
재배품종들과 야생 액세션들 간의 높은 FST (95% 분위수)를 갖는 유전자를 파악했다.
이로 인해 1,908개의 유전자 세트가 생성되었는데,
GO 카테고리 ' cellular response to nitrogen starvation (窒素饑餓에 대한 세포 반응)',
'regulation of innate immune response (선천적 면역 반응 조절)’ 그리고
'regulation of defense response (방어 반응 조절)'(Supplementary Table 11)에 대해 강화되고,
Coffea의 질소 흡수에 중요한 [63] 암모늄 수송체 AMT1과 AMT2의 동족체(상동체, homologs)를 포함할 뿐만 아니라 ;
살리실산 신호 전달(salicylic acid signaling) 및 전신 획득 저항(systemic acquired resistance)에 필요한,
살리실산 수용체 NONEXPRESSER OF PR GENES 1(NPR1)의 동족체 [64]; 뿐만 아니라
다른 계(kingdoms)로부터의 병원체 반응기(effector)에 의해 집중적으로 표적화되는 허브로 이전에 식별된 Arabidopsis LSU2 유전자의 상동체 [65]도 포함한다.
재배품종들 사이에서 공유되는 영향이 큰 비유사 돌연변이(nonsynonymous mutations)가 많은 유전자(돌연변이가 있는 개체들이 40% 이상)에 초점을 맞춘 두 번째 스크린에서는, 단 하나의 GO 카테고리인 ‘defense response (방어 반응)’에 대해 상당히 강화된 556개 유전자 목록을 생성했다 (Supplementary Table 12).
이 카테고리의 22개 유전자 중 16개는 NB-ARC domain-containing resistance (R) 유전자였고,그리고 2개는 leucine-rich repeat (LRR) defense gene 계열의 구성원이었다.
면역 관련 반응들에서의 높은 다양성은 식물 세계에서의 가능한 병원체 저항 메커니즘 (pathogen resistance mechanism) 중 하나이며 [66], 따라서 감소된 다양성(reduced diversity)은 현대 아라비카 재배품종의 면역력을 손상해왔을 수도 있다.
아라비카 서브 게놈들과 그들의 이배체 조상들(progenitors) 사이의 높은 보존 수준은 자발적 종간 교잡화 이벤트들(spontaneous interspecific hybridization events)을 촉진했을 수 있다.
이는 H. vastatrix에 대한 저항성을 갖는 자발적인 Robusta × Arabica hybrid인 Timor 하이브리드의 경우이다 [27].
우리의 샘플 세트에는 아라비카와의 여교배(backcrossing)를 통해 얻은 오리지날 Timor 하이브리드의 후손 5개가 포함되어 있다.
기대한 대로, 그 하이브리드화는 재배품종들과 하이브리드들을 비교할 때, subEE(FST = 0.0897)에서 보다 훨씬 높은 수준의 뉴클레오티드 분기(nucleotide divergence) (FST = 0.185)로 subCC에서 더 심오하게 영향을 미쳤다.
야생 개체군들로부터의 분기는 subCC의 경우 FST = 0.254, subEE의 경우 FST = 0.138로 훨씬 더 컸으며, 이는 유전자 이입이 subCC 내에서 거의 독점적으로 발생했음을 나타낸다.
Timor 하이브리드에서, df 통계 [61]로 발견된 영역들은 FST scans를 사용하여 파악된 유전자이입 유전자좌들과 크게 겹치며 (Fig. 4a), 최근 혼성화(recent hybridization)를 반영하고 게놈의 7~11%를 차지하는 큰 블록에서 발견되었다 (Fig. 4a and Extended Data Fig. 8).
트랜스포존 삽입 다형성(Transposon insertion polymorphisms, TIP)도
유전자이입된 영역들(introgressed regions)과 겹쳐서 (Gypsy P = 0.0002, Copia P = 0.035, Fisher exact test),
Robusta로부터 최근에 유래되었음을 확인했다 (Fig. 4b).
유전자이입된 영역들(introgressed regions)은
더 높은 서브 게놈 분별(introgressed regions)의 영역들과 겹쳤는데 (P = 0.001873, Supplementary Table 13),
이는 아마도 subCC와 Robusta 간의 이종 기원 재결합 (heterologous recombination)으로 인해
불평등한 교차(unequal crossing-over)가 발생했기 때문일 수 있다.

모든 Timor 하이브리드 계통들이 공유하는 유전자이입 영역(an introgressed region)은 염색체 4에서 분명했다 (Fig. 4a).
우리는 모든 하이브리드들이 공유하는 233개의 유전자 세트를 확인했다 (Supplementary Table 14).
이 세트는 염색체 4, subCC에 3개의 동일 위치에서 함께 발생하는 직렬로 복제된 저항 관련 유전자 블록 (colocalized tandemly duplicated blocks of resistance-related genes)의 멤버들을 포함하고 있으며, 재배품종들과 유전자이입 계통들 사이에 높은 FST 값을 보여주었다.
5개 유전자의 직렬 배열(tandem array)은 몇 가지 병원체들에 대한 多面發現性 抵抗(pleiotropic resistance)을 부여하는 NOD-유사 수용체 저항 유전자좌인 Arabidopsis (애기장대) RPP8의 상동체들이었다 [67,68].
RPP8은
Arabidopsis (애기장대)에서만 엄청난 양의 변이(variation)를 보여주며,
염색체 내 유전자 전환(intrachromosomal gene conversion)과 균형 선택(balancing selection)이 결합되어
예외적인 다양성에 기여한다.
동일한 subCC 영역에는 저항 단백질(resistance proteins)을 표적으로 하는 방어 반응의 음성 조절자 (a negative regulator)인 CONSTITUTIVE EXPRESSER OF PRGENES 1(CPR1)의 10개 상동체들(homologs)의 직렬 배열도 포함되어 있다 [70,71].
마지막으로, 우리는 잎 녹병 10 질병-저항성 유전자좌 수용체-유사 단백질 키나제 (Leaf rust 10 disease-resistance locus receptor-like protein kinases) (LRK10L)를 인코딩하는 세 개의 복제들(three duplicates)을 확인했다.
LRK10L은 식물 전체에 널리 퍼져 있는 유전자군이다.
밀의 잎 녹병 저항성에 기여하는 위치에서 단백질 키나아제(protein kinase)로 처음 확인되었으며 [72],
그것들은 다양한 생물학적 및 비생물적 스트레스 동안 상향 조절되는 것으로 밝혀졌고 [73],
줄무늬 녹병 곰팡이(stripe rust fungus) [73] 및 흰가루병(powdery mildew) [74]에 대한 밀 과민성 저항 반응(wheat hypersensitive resistance response)의 양성 조절자(positive regulators)로 확인되었다.
야생 개체가 아닌, 재배되는 개체와 유전자 이입된 개체들 사이의 높은 FST 값들(Fig. 4b)은
야생 개체군이 대립유전자 비대칭(allelic asymmetries)의 원인이 될 수 없음을 나타낸다.
뉴클레오티드 다양성(Nucleotide diversities)은 이 점을 더욱 잘 보여준다; 일부 유전자들은 야생 개체들에서 더 낮은 뉴클레오티드 다양성을 보여 주며, 이는 이러한 유전자들이 선택적 스윕(selective sweeps)을 경험했음을 시사한다.
잎녹병 저항성과 관련된 후보 유전자를 더욱 좁히기 위해 우리는 H. 바스타트릭스 접종 후 감수성 및 저항성 수탁자로부터의 비교 유전자 발현 데이터를 재분석했다 [75].
이 분석에서는 723개의 차별적으로 발현된 유전자가 확인되었으며, 그 중 대부분은 방어 반응과 관련이 있었다 (Fig. 4b 및 Supplementary Table 14 and 15).
높은 FST 값, 뉴클레오티드 다양성 및 차등 발현 데이터의 조합은 이 유전자좌에서 여러 강력한 후보 유전자(RPP8 1개, CPR1 6개 및 LRK10L 1개)를 강조한다.
가장 중요한 농산물들 중 하나의 분자적 육종을 위한 게놈 자원을 제공하는 것 이외에도, 우리의 Arabica, Robusta 그리고 Eugenioides 게놈들은 2개의 가까이 관계된 종들로부터 나온 최근에 형성된 이질배수체의 게놈 진화에 대한 독특한 윈도우를 제공해준다.
우리의 Arabica 데이터는
이질배수성(allopolyploidy)에 의해 유도된 게놈 쇼크(genomic shock)를 시사하지는 않았지만,
대신에 유일한 더 높은 LTR 트랜스포존 전환속도(LTR transposon turnover rate)을 제시했다.
게놈 분별률(Genome fractionation rates)은
이질배수체 사건(the allopolyploidy event) 전후에 기본적으로 변경되지 않은 상태로 유지되었다.
마찬가지로, 유전자 발현에서 전체적인 서브게놈 우세는 관찰되지 않았지만, 오히려 다른 최근 배수체들에서와 같이 모자이크 타입 패턴이 관찰되어 [10,44], 개별 유전자 계열 구성원의 발현에 영향을 미쳤다.
그러나 8배체(octoploid) 딸기와 유사하게 [8],
우리는 subCC를 선호하는 편향된 HE의 관점에서 게놈 우성(genome dominance)을 발견했다.
Robusta는 Coffea 屬에서 가장 넓은 지리적 범위 중 하나를 가지고 있는 반면,
Eugenioides는 범위가 더 제한적이므로 이 편향된(biased) HE는 적응력이 있을 수 있다.
이 가설은 HE 유전자좌의 위치 주파수 스펙트럼(the site frequency spectrum)에 의해 뒷받침되었으며, 방향 선택의 징후(signs of directional selection)를 보여준다 (Extended Data Fig. 3).
흥미롭게도, 전이 가능한 삽입 다형성(transposable insertion polymorphisms)은
직렬 유전자 복제 (tandem gene duplications) 및 생합성 유전자 (biosynthetic gene) 클러스터들과 상당히 겹쳐서
클러스터 진화에서 그들의 가능한 역할을 암시한다.
아라비카 커피와 같은 다년생 종의 재배화(domestication)는
야생 숲으로부터 뛰어난 유전자형의 선택 (selection of outstanding genotypes from wild forests),
클론 번식 및 경작 (clonal propagation and cultivation),
육종 및 다양화 (breeding and diversification)의 3단계로 구성된다는 점에서
일년생 작물(annual crops)의 경우와 현저히 다르다 [76].
다년생 작물(perennial crop)일 뿐만 아니라 아라비카는
주로 자화수분 이질배수체(autogamous allopolyploid)로서 독보적인 분류에 속한다.
우리는 여기에서
여러 재배화 전 병목 현상들(multiple pre-domestication bottlenecks)로 인해
야생 액세션들 사이에서 유전적 다양성이 이미 매우 낮았으며,
인간이 재배하기 위해 선택한 유전자형(고대 재배 에티오피아 랜드레이스들과 최근 Geisha 재배품종 모두)이
이미 서로 다른 계통들 간에 어느정도 혼합되어 있음을 보여준다.
재배열된 액세션들은
the Great Rift Valley의 동부과 서부로 지리적 분리를 나타냈으며,
재배되는 커피 변종들은 모두 동부 개체군과 함께 처하게 되었다.
이러한 혼합은 많은 유실작물들을 육종하는 데 큰 역할을 했으며, 비-배수체 타화수분성 다년생 리치 (non-polyploid allogamous perennial lychee)는 가장 극단적인 경우 중 하나이다 [58].
아라비카의 보편적인 자화수분(autogamy)은,
야생에서 겪었던 여러 유전적 병목 현상이 결합되어,
해로운 대립 유전자들(deleterious alleles)을 선택적으로 제거했을 수 있으며,
이는 그것의 경작 중 발생한 단일 식물 병목 현상 (single-plant bottlenecks)에서
살아남을 수 있는 종의 능력을 설명한다.
해로운 대립유전자들로부터 보호하는 추가적인 요소는
아마도 아라비카의 이질배수성 (allopolyploidy) 자체였을 것이며,
이는 일정 수준의 잡종강세(heterosis)를 제공했다 [77].
그러나, 재배되는 아라비카와 현대의 야생 아라비카 모두의 좁은 유전적 기반은
야생 유전자 풀의 다양성을 활용한 육종에 장애물일 뿐만 아니라 주요 약점을 구성한다.
한편, CC 및 EE 서브게놈과 Robusta 및 Eugenioides 조상들의 서브게놈의
광범위한 공선성 (extensive collinearity)은,
역사적으로 Timor 자생 하이브리드에서 이미 발생한 것처럼,
이들 종으로부터 흥미로운 특성의 유전자 이입(introgression)을 촉진할 가능성이 높다.
본 연구에서 제공된 3종의 고품질 게놈 서열들은,
커피 잎 녹병에 대한 저항성을 부여하는 게놈 영역의 식별과 함께,
우수한 적응성과 병원체 저항성을 갖춘 새로운 아라비카 품종 육종을 위한 초석을 구성한다.





- 3가지 Coffea 종들에 대해, 게놈 DNA가 잎 조직들로부터 추출되었다.
- Qiagen kit가 Illumina sequencing을 위한 DNA 추출에 사용되었다.
- Illumina short reads 및 PacBio 20-kilobase (kb) libraries가 제조업체의 지시에 따라 마련되었다.
- 시퀀싱은 short reads의 경우 HiSeq2000 기기에서, long reads의 경우 PacBio RSII 플랫폼에서 수행했다 (스펙은 Supplementary Table 16에 나와 있음).
- HiFi reads 생성을 위해, 원심분리에 의한 핵 정제(nuclei purification) 후 용해(lysis), 페놀-클로로포름 추출(phenol–chloroform) 및 이소프로판올 침전(isopropanol precipitation)을 거쳐 C. arabica 잎 조직에서 DNA를 추출했다.
- Megaruptor 3를 사용하여 DNA를 20kb로 단편화했다.
- SMRTbell 라이브러리는 Sequel IIe 플랫폼의 단일 SMRTcell에서 시퀀싱되었다.
- 39개의 야생 및 재배 C. arabica 액세션들의 재서열화 (resequencing)를 위해,
라이브러리들이 제조업체의 지침에 따라 KAPA HyperPrep Kits (Roche)을 사용하여 마련되었고,그리고
Illumina HiSeq2500 장비에서 ~40× 적용 범위까지 paired-end (2 x 125) 시퀀싱했다. - Linnaean 식물표본관 샘플은 Ion Torrent 기술을 사용하여 46× coverage로 배열되었다.

- C. canephora에 대한 Contig-level 어셈블리는 MHAP79를 사용하여 얻었으며,
이전에 생성된 BAC-end sequences와 454개의 paired-end sequences를 사용하여 스캐폴딩되었다 (scaffolded) [33]. - C. eugenioides와 C. arabica는 모두 Falcon80으로 어셈블리되었으며,
C. arabica는 이후 Falcon_unzip을 사용하여 단계적으로 분류되었다. - 세 가지 게놈들 모두 Illumina short reads를 사용하여 Pilon [81]으로 오류 수정되었다 (Supplementary Section 2.2).
- C. canephora와 C. arabica는 Dovetail Hi-C 기술을 사용하여 유사염색체(pseudochromosomes)에 추가로 스캐폴딩되었다 (scaffolded).
- C. eugenioides의 경우, 어셈블리 연속성(the assembly contiguity)의 추가 개선을 위해 더 이상 재료를 얻을 수 없었으며, 그리고 그 어셈블리는 C. canephora를 레퍼런스로 사용하여 의사 분자들(pseudomolecules)로 스캐폴딩되었다.
- 스캐폴드들의 틈들(gaps)을 PBJelly로 채웠고 [82], 그 후 Illumina shotgun sequenced genomic DNA와 RNA sequencing (RNA-seq) reads (판독)을 사용하여 Pilon으로 6회 더 폴리싱(polishing)을 수행했다.
- C. canephora에 대한 결과로 나온 염색체 어셈블리는 프로젝트 중에 생성된 초고밀도 연결 맵 (linkage map) [83]을 사용하여 확인 및 수정되었다.
- C. arabica 어셈블리의 품질을 더욱 향상시키기 위해, Bionano genome maps가 생성되었다.
- C. arabica HiFi 어셈블리는 hifiasm v.0.16.1 (ref. 84)을 사용하여 수행되었으며,
Dovetail 기술 및 ALLHiC [85] 파이프라인의 Hi-C 데이터를 사용하여 스캐폴딩이 수행되었다. - 어셈블리의 최종 품질 검사와 수동 조정은 3d-DNA [86] 및 juicebox [87]를 사용하여 수행되었다.
- 다양한 어셈블리의 완전성은 eudicots_odb10 데이터베이스(2,326개 유전자, 표 1)와 함께 BUSCO v.5.2.2(참조 35)를 사용하여 평가되었다.
- CoGeBLAST를 사용하여 염색체 전체에서 텔로미어 반복(Telomeric repeats)을 검색했다 [88].
- C. arabica의 두 서브게놈의 위상 조정(phasing)을 평가하기 위해,
CoGe에서 synonymous nucleotide substition (Ks) 값을 얻었고 [89],
C. arabica와 두 이배체 외부 그룹인 C. canephora 및 C. eugenioides 각각을 R에서 스크립트를 사용하여 비교했다.
- 레퍼런스 유전자 지도가 콩고 그룹 유전자형 (BP409)과 콩고 × 기니 하이브리드 부모(Q121) 사이의 교배로부터 구성되었다.
- 그 분리 집단(segregating population)은 93개의 F1 개체들로 구성되었다 [90].
- Nestlé Research의 Illumina HiSeq2000 플랫폼을 사용하여 부모는 60×로, 자손은 20× 범위로 시퀀싱되었다.
- FastQC를 사용한 품질 관리 및 Trimmomatic v.0.36(참조 91)을 사용한 트리밍(trimming)에 이어
BWA-MEM v.0.7.15 (ref. 92)를 사용하여 C. canephora 참조 어셈블리에 대해 판독 값(reads)을 맵핑했다. - linkage mapping은 Lep-MAP3 (ref. 83)을 사용하여 수행되었다.
- 분리 왜곡 인식 모델 (segregation distortion aware model)에서 확률 점수(odds score) 18의 로그를 사용하여
마커를 부계 및 모계 연결 그룹으로 클러스터링했다. - 두 개의 부모 맵을 결합하고 충돌을 해결하고 일배체형 대립유전자들(haplotype alleles)를 식별하는 어셈블리의 최종 큐레이션(curation)이 수작업적으로 수행되었다.
- EDTA [93]가 C. canephora, C. eugenioides, 그리고 C. arabica 서브게놈들에서 TE를 새롭게 식별하는 데 사용되었다.
- Inpactor2 (ref 94)는 세 가지 게놈에서 full-length LTR retrotransposons을 복구하고
이를 계통 수준(lineage level)에서 분류하는 데 사용되었다. - EDTA 및 Inpactor2 라이브러리들은 cd-hit [95]를 사용하여 병합 및 클러스터링되었다.
- 중첩된 예측과 잘못된 예측을 제거하기 위해 클러스터를 수동으로 검사했다.
- 큐레이션 후에는 Repeat Masker를 사용하여 (디폴트 파라미터), 주석(annotation)에 라이브러리들을 사용했다.
- Length >200 base pairs (bp)인 주석(Annotations)은 유지되었다.
- LTR retrotransposon insertions의 타이밍은 Orozco-Arias et al. [97]과 유사하게,
Inpactor2에 의해 복구된 개별 서열들과 average base substitution rate (평균 염기 치환율) 1.3 × 10-8 (ref 96)을 사용하여 세 가지 게놈들 에서 연구되었다.

- 새로운 유전자 예측(de novo gene prediction) 지원을 위해 RNA-seq 및 IsoSeq reads가 생성되었다.
- MAKER-P pipeline [98]이
여러 de novo gene callers를 IsoSeq 및 short-read RNA-seq으로부터의 접합 정보(junction information)와 결합하는 데 사용되었다. - Annotation Edit Distance score가 0.5 미만인 고-증거 유전자 모델(High-evidence gene models)이
주석(annotation)을 위해 선택되었다. - C. arabica HiFi 어셈블리의 경우,
먼저 GeMoMa v.1.9 (ref. 99)를 사용하여 CC, CE 및 이전 CA assembly로부터
주석(annotations)을 전송한 다음 결합했다. - 커피 플레이버와 관련된 모든 관심 유전자들은 수작업 검사와 유전자 모델 큐레이션을 거쳤다.
- 주석에 이어 CC, CE 및 CA predicted transcriptomes (예측 전사체)에 대해 BUSCO completeness scores (완전성 점수)를 평가했다.
- Terpene synthases (TPS), N-methyltransferases (NMT) 및 fatty acid desaturase 2 (FAD2)를 인코딩하는
세 가지 유전자 계열들(gene families)을 추가로 특성분석하고,
이전에 발표된 발현 데이터 [100]를 사용하여 이질배수체 (allopolyploid)에서 추가 유전자 복사본(extra gene copies)의 존재 영향을 조사하는 데 사용했다. - 여기에 제시된 발현 데이터는
로그 스케일링 (log10(x + 1 × 10−4), 여기서 x는 STARaligner101의 TPM count)을 사용하여 정규화된
TPM (transcripts per million) 카운트이다. - Leaf rust 차등 발현 분석(differential expression analysis)을 위해,
이전에 발표된 RNA-seq 데이터 [75]는 STARaligner를 사용하여
C. arabica HiFi assembly 상에서 그 reads를 맵핑함으로써 재분석되었다. - H. vastatrix를 접종한 후
Timor hybrid 대 취약한 Caturra 액세션에서의 차별적 발현(Differential expression)은
R의 DEseq2 (ref. 102)로 분석되었다. - FDR (False discovery rate) 조정은 Benjamini-Hochberg method을 사용하여 수행되었다;
adjusted P value < 0.05는 통계적으로 유의한 것으로 간주되었다.

- Synteny information이 the CoGe platform에서의 SynMap tool을 사용하여 얻어졌다 [88,89].
- 斷裂 (조각화, fractionation)나 다른 유전자 손실(gene loss) 때문에,
다른 게놈에서 그들의 카운터파트를 잃은, 각 게놈에서의 유전자 쌍들 뿐만 아니라
singleton genes도 포함하여,
Synteny blocks 내의 유전자들만 고려되었다. - 우리는 C. arabica의 게놈 진화 과정 동안에
중복 게놈들(duplicate genomes)을 생성하는 세 가지 사건들, 즉
핵심 진정 쌍떡잎식물(core eudicots)의 기원에서의 감마 삼중화 (gamma triplication ),
CC/CE divergence (분기)에 관련된 종분화(speciation), 그리고
이질배수체화 이벤트(allotetraploidization event)에 대해,
R function geom_density에 의해 계산되는 ‘peaks’ method를 사용했다. [103].

- CE, CC, subCC, 그리고 subEE 간의 Syntenic genes (신테닉 유전자들, 동염색체 유전자들)는
CoGe 플랫폼의 SynMap 툴를 사용하여 식별되었다. - 대립유전자 편향(allele biases)의 식별은
결합된 CE 및 CC 어셈블리들에 대해 C. arabica short-read sequencing 데이터를
BWA-MEM [92]을 사용하여 매핑하고,
bedtools을 사용하여 신테닉 유전자들에 대한 염색체들에 걸친 시퀀싱 커버리지를 계산하여 수행되었다. - Custom R scripts를 사용하여
염색체 전체의 차등 커버리지(Differential coverage across the chromosomes)를 시각화했다. - 노이즈를 줄이기 위해
10개 유전자의 슬라이딩 윈도우를 사용하여 염색체들을 따라 average coverage를 계산했다. - 대립유전자 밸런스(allele balance)는 A = 4 × ((CC/(CC + EE)) - 0.5)로 계산되었으며,
여기서 CC와 EE는 각각 subCC 및 subEE syntelog coverages이다.
(※ syntelog ⇒ 유전자 세트가 동일한 조상 게놈 영역에서 파생된 유전자 상동성의 특별한 경우) - 대립유전자 밸런스 (allele balance)가
<-1.5 or >1.5 (-1.5보다 더 작거나, 1.5보다 더 큰 밸런스)이면,
각각 EE 또는 CC에 대해 동형접합적(homozygous)인 것으로 간주되었으며,
<0.5 and >-0.5는 동일한 것(equal)으로 간주되었다 (⇒ 동형 유전자를 가지고 있는지, 동일한 것인지).
- FastQC [104]를 사용한 품질 관리에 이어,
Illumina short reads는
Trimmomatic v.0.36 (ref. 91)을 사용하여 트리밍 되었으며,
BWA-MEM v.0.7.16a-r1181 (ref. 105)을 사용하여 C. arabica 레퍼런스 어셈블리에 매핑 되었다. - Linnaean 샘플의 경우,
MapDamage v.2.0.8 (ref. 106)의 degraded DNA analysis에서 권장되는 프로토콜에 따라 reads가 처리되었다. - SNP calling에는 GATK(v.3.8.0) pipeline이 사용되었다.
- Picard v.2.0.1을 사용하여 중복 항목들(duplicates)을 표시하고 제거했으며,
HaplotypeCaller (GATK)를 사용하여
GVCF 파일에 유전자형 尤度(genotype likelihoods)가 호출되었다(called). - 이배체 조상(diploid progenitors)의 경우, 종간 비교를 가능케 하기 위해,
염색체 제로(chromosome zero), 즉 두 mappings 모두에서 의사 분자(pseudomolecules)로 조립되지 않은 콘티그(contigs)를 포함하여, 각 서브게놈들에 있어서 개별적으로 매핑이 수행되었다. - Joint calling (공동 호출)이 GenotypeGVCF (GATK) [107]을 사용하여 수행되었으며,
snpEff v.4.3t를 사용하여 SNP [108]의 영향을 평가했다. - Cross-species mappings가 있는 영역들을 제거하기 위해, 우리는
di-haploid (이반수체) ET-39 시퀀싱 데이터를 아라비카 레퍼런스 게놈에 매핑할 때
이형접합성(heterozygous)이라고 불리는 SNP를 제거했다. - Genome-wide nucleotide diversity (게놈 전체의 뉴클레오티드 다양성)은
vcftools v.0.1.17 (ref. 109)을 사용하여
10kb step size기의 100kb sliding windows에서 pi values의 평균을 계산함으로써 계산되었다. - 마찬가지로 genome-wide Tajima’s D는
window size가 100kb인 Tajima’s D 값들의 평균으로부터 계산되었다. - PCA는 Plink v.1.90 (ref. 110)을 사용하여 실행되었다.
- ADMIXTURE v.1.3.0 (ref. 111)은
repeat regions의 변이들(variants)가 필터드 아웃되고
outgroup species (이배체 Coffea 종)이 제외되는 SNP 데이터에 대해 실행되었다. - 최대 10% missing values (결측값)(--geno 0.1)을 허용하면서,
ADMIXTURE 매뉴얼의 권장 사항(--indep-pairwise 50 10 0.1)에 따라,
SNP를 linkage disequilibrium (LD)에 대해 필터링했다. - Admixture analysis (혼합물 분석)는
10배 교차 검증(tenfold cross-validation)을 사용하여 실행했다. - 교차 검증 점수(cross-validation score)가 가장 낮은 솔루션이 최상의 솔루션으로 선택되었다.
- Nonsynonymous nucleotide diversity π0, 그리고 neutral, intergenic πs는
ref. 8과 유사하게 PiNSiR R package (https://github.com/jsalojar/PiNSiR) 및
ANGSD v.0.933 (ref. 112)을 사용하여 계산되었다.

- C. arabica의 736개 PstI GBS libraries로부터의 read data는
SRA repository (저장소) (bioproject PRJNA554647)에서 다운로드 되었다. - 샘플은 Illumina HiSeq2000 기기에서 시퀀싱된 100-bp single-end reads이다.
- 트리밍 및 품질 필터링 후 데이터는,
BWA v.0.7.17 (ref. 105)의 디폴트 설정으로 BWA-MEM algorithm을 사용하여
C. arabica의 레퍼런스 게놈 서열에 매핑되었다. - SNP는 GATK v.3.7 (ref. 107)의 Unified Genotyper를 사용하여 called되었다.

- F3 통계량을 계산하는 데 Admixtools package [113]가 사용되었으며,
얻은 P values는 Salojärvi et al. [114]이 개발한 절차를 사용하여 FDR 보정 (correction)을 거쳤다. - Z-scores를 P values로 변환한 후,
Benjamini-Hochberg correction을 사용하여 FDR 보정을 수행한 다음,
다시 Z-scores로 변환했다.

- SNPs를 반복 영역(repetitive regions)에 대해 필터링한 다음,
LD > 0.4 및
missing values가 >40%인 유전자좌들(loci), 뿐만 아니라
마이너 대립유전자 출현율 (minor allele prevalence) <10%인 것들도 필터링했다. - 선택된 사이트들에서 얻은 fasta 파일은
30개의 시작 트리(starting trees)와 1,000개의 부트스트랩 샘플(bootstrap samples)을 사용하여,
-T 30 -m GTRGAMMA model로 하여 RAxML에 입력되었다 [115].

- 각 개별 개체에 대해, reads가 full CA 레퍼런스 어셈블리에 대해 매핑되었다.
- 그런 다음 bcftools와,
<8× 또는 >100× coverage를 가진 영역들을 사용하여 indels에 대해 이 매핑을 필터링했다. - 필터링 후,
얻어진 PSMC (pairwise sequentially Markovian coalescent) fastq 파일이
subCE 및 subCC 특정 부분들로 분할되었으며,
PSMC 데모그래피가 표준 파라미터 세팅 (-N25 -t15 -r5)을 사용하여 추정되었다 [116]. - 그 다음에 그 추론된 히스토리를 R package와 ggplot2 package를 사용하여 시각화하였다.

- 조상 상태(ancestral state)는
각각의 서브게놈들과 할당되지 않은 콘티그들(unassigned contigs)에 대해 매핑 된
C. canephora (BUD15, Q121) 및 C. eugenioides (BU-A, DA56)의
각 이배체 (diploid) 커피 종의 두 대표들의 reads로부터 추론되었다. - 그 후, -doFasta 2 및 -doCounts 1 옵션으로,
ANGSD v.0.933 (ref. 112)을 사용하여
조상 대립유전자(ancestral allele)를 추론하기 위해 다수결(majority vote)이 수행되었다. - 그런 다음 VCF 파일의 SNP calls가 bcftools + fixref [117]를 사용하여
ancestral states로 전환되었다 (flipped to).
- SMC++의 입력 데이터는
ancestral state가 래퍼런스로 사용되었고(위 참조) 그리고 반복 영역(repeat regions)의 SNP가 필터링된 VCF 파일로 구성되었다. - 재배품종 집단(cultivar population)의 경우,
Bourbon 및 Typica 계통(lineages)의 대표들이 포함되었다 (TIP1, Bourbon, Mundo Novo, BMJM, Moka, Rubi, Topazio, Bourbon pointu, Catuai99, BB1, Electa, JK1, Guatemalense, Amsterdam);
게이샤 (Geisha)는 혈통(pedigree)을 알 수 없기 때문에 분석에서 제외되었다. - SMC++ 파라미터 선택은
SMC++ v.1.15.3 (ref. 51)에 구현된 3중 교차 검증(threefold cross-validation) (smc++ cv)을 사용하여 수행되었다.

- 친족 분석(kinship analysis) 전에,
이배체 종들(diploid species)을 SNP 파일에서 제거하고,
KING software v.2.2.5 (--kinship option)를 사용하여 친족 관계를 추정했다 [59]. - 결과는 Keynote를 사용하여 각 서브게놈에 대해 개별적으로 시각화되었다.

- Orientagraph v.1.0 (ref. 60)이
TreeMix [118]에서 권장된 바와 같이, 연결에 대한 필터링(filtering for linkage)을 수행하여
개발자 권장사항에 따라, 각 서브게놈에 대해 개별적으로 실행되었다. - PopGenome R package가 d_f statistics를 계산하는 데 사용되었다 [61].
- subCE introgression (유전자이입)의 경우,
BUD15는 아웃그룹(outgroup)으로,
DA56은 유전자 이입 소스(source of introgression)로, 그리고
E383은 혼합되지 않은 야생 대표(nonadmixed wild representative)로 사용되었다. - subCC의 경우
DA56가 아웃그룹으로 사용되었고,
BUD15가 이입 소스로 사용되었다. - 유전자 이입의 중요성을 평가하기 위해,
weighted jackknife를 사용하여 20kb 비중첩 창(nonoverlapping windows)에서 통계량을 계산했다. - 결과는 R을 사용하여 시각화되었다.

- FastSimCoal v.2.6가 개체군 시뮬레이션 (population simulations)을 위해 사용되었다. [54]
- Site frequency spectrum은,
필터 아웃된 야생 개체들(wild individuals)과
반복영역들(repetitive regions)이 포함된 VCF 파일을 가지고
ANGSD112를 사용하여 계산되었다. - Ancestral states는 위에서 설명된 대로 추정되었다.
- 각 모델들에 있어서, 100개의 파라미터 파일들이 시뮬레이트되었다.
- 각 파라미터 파일들에 있어서, 1백만번의 시뮬레이션이 실행되었다; monomorphic sites는 사용되지 않았다.
- 파리미터들에 대한 최대복합우도 추정(Maximum composite likelihood estimation)이
40회 기대-조건 최대화 아이터레이션(expectation-conditional maximization iterations)으로 수행되었다.

- vcftools를 사용하여
각 유전자 주석(gene annotation)과 2kb 측면 영역(flanking regions)에 대해
야생 개체와 재배 개체 간의 Site-wise FST values을 계산했다 [109]. - 그런 다음 R 패키지를 사용하여 각 유전자 모델에 대해 평균 FST 값을 계산했다.

- 우리는 discordant mapping pair approach (불일치 매핑 쌍 접근법)을 사용하여
TIP_finder [119]를 이용해서 short-read whole-genome resequencing data의 분석을 통해
LTR 레트로트랜스포존 삽입 (retrotransposon insertions)을 연구했다.
- 생합성 유전자 클러스터 (Biosynthetic gene clusters)는
디폴트 분석 프로토콜에 따라
Plantismash 웹 서버(http://plantismash.secondarymetabolites.org/)로 식별되었다 [120].

- 다양한 유전자 세트들 간의 오버랩들의 통계적 유의성(Statistical significance)은
R의 Fisher exact test를 사용하여 평가되었다. - 유전자 세트 농축 (Gene set enrichments)은
먼저 각 유전자를 가장 가까운 Arabidopsis homolog (애기장대 동족체)의 GO category에 할당하여
수행되었다 (E-value threshold 1 × 10−5 사용). - 농축 테스트(Tests for enrichment)는 goatools를 사용하여 수행되었다 [121].
- Bonferroni-corrected P value 0.05가 유의성에 대한 임계값(threshold)으로 사용되었다.
- 대립유전자 밸런스(allele balance)에 대한 테스트는
chi-squared test를 사용하여 수행되었다; 각 테스트에는 d.f.=1이었다.

- 연구 디자인에 관한 추가적인 정보는
본 아티클에 링크된 Nature Portfolio Reporting Summary에서 구할 수 있다.

커피 게놈 어셈블리들은 CoGe (https://genomevolution.org/)에서 구할 수 있다 :
C. canephora: 50947;
C. eugenioides: 67315; and
C. arabica: 66663 (Pacbio HiFi) and 53628 (Pacbio).
게놈 데이터는 또한 ORCAE에서도 구할 수 있다.
(https://bioinformatics.psb.ugent.be/orcae/overview/Coara and
https://bioinformatics.psb.ugent.be/gdb/coffea_arabica/).
모든 시퀀싱 데이터는 NCBI under bioproject ID PRJNA698600에서 구할 수 있으며,
우리의 어셈블리들은 거기서 다음과 같은 것으로 액세션되어 있다.
JAZHSI000000000.1, JAZHGF000000000.1, JAZHGH000000000.1 and JAZHGG000000000.1.
Genotyping data (VCF files)와 syntenic alignments는 Data Dryad에서 구할 수 있다:
https://doi.org/10.5061/dryad.qnk98sfpt.

Neutral and deleterious nucleotide diversities (PiNSiR) 계산을 위한 R scripts는
Zenodo에서 제공되어 있다 [122]. (https://zenodo.org/doi/10.5281/zenodo.5136526).



'Coffee Genetics' 카테고리의 다른 글
| 1세대 DNA 시퀀싱 (22) | 2024.07.01 |
|---|---|
| 식물 세포, 핵, DNA, 유전자, 게놈 (1) | 2024.06.29 |
| 아라비카 커피 기원의 싱글 다배체화 사건이 극히 낮은 유전적 변이의 원인 (0) | 2024.06.05 |
| SSR Genotyping (13) | 2024.05.14 |
| Molecular characterisation and origin of the Coffea arabica L. genome (25) | 2024.05.08 |




댓글