

1. Introduction
- 작물 다양성(Crop diversity)은 ⇒ 지속 가능한 농업 생산의 핵심.
- 이는 오늘날의 생산을 뒷받침하고 급변하는 세계에 맞서
내일의 지속적인 공급을 보장하는데 필요한 원자재를 제공한다. - 種의 유전적 다양성(genetic diversity)이 넓을수록
⇒ 질병, 해충, 날씨 변화와 같은 문제에 대처할 수 있는 회복력(capacity for resilience)이 더 커진다. - 전 세계적으로
커피 생산이 ⇒ 게놈 및 표현형 다양성이 감소된 (reduced genomic and phenotypic diversity)
수의 재배품종 (cultivars)에 의존하고 있는 반면,
야생 커피나무는 ⇒ 엄청난 유전적 다양성을 보유하고 있다. - 커피가 감당할 수 있는 작물로 생존할 수 있도록 보장하는 열쇠는
커피 생식질 컬렉션들에서 발견되는 유전적 변이(genetic variation)에 달려 있다는 것이 이제 일반적으로 인정된다. - 이러한 변이는
⇒ 불안정한 기후와 식물 질병들의 영향을 완화하는데 도움이 될 뿐만 아니라
⇒ 커피 씨앗에 존재하는 건강 관련 화학 물질들을 바꾸는데 도움이 될 것이다. - 이 식물 그룹이 나타내는 높은 적응 능력과 관련하여 많은 수의 커피 종들이 기술되었다.
- 이들 종의 특성과 분포는
표현형 및 생물학적 특성과 틈새 점유 측면에서 매우 다양하다. - 더욱이, 거의 모든 커피 종은
잠재적으로 교배하여, 비교적 번식력이 있는 종간 교잡종(interspecific hybrids)을 생산할 수 있으며,
이를 통해 야생 커피의 특성이 Coffea arabica 또는 C. canephora 재배품종으로 전달될 수 있다. - ‘오믹스(omics)' 테크놀로지의 급속한 발전은
작물 종에 대한 새로운 데이터 세트를 생성할 수 있는 기회를 제공한다. - 게놈 및 기능적 오믹스 데이터를 유전적 및 표현형 정보 (genetic and phenotypic information)와 통합하면
중요한 농업적 표현형을 담당하는 유전자 및 경로가 식별된다. - 하지만 이 거대한 유전자원을 최대한 활용하기 위해서는 커피 다양성에 대한 기본 지식이 필요하다.
특히, 관찰되는 유전적, 게놈적 다양성의 분자적 기초를 아는 것뿐만 아니라
커피 종 간의 다양화 메커니즘을 아는 것이 필요하다. - 이를 위해 지난 10년 동안 여러 가지 다양성 및 게놈 연구가 시작되었다.
- 아직 불완전하기는 하지만
커피 종 간의 계통발생적 관계와 커피 종 다양화의 분자 기반과 관련하여 상당한 진전이 이루어졌다.
2. Coffee diversity
2.1 Taxonomy
- 식물학적 관점에서 커피나무는,
⇒ 속씨식물에서 네 번째로 큰 科인
꼭두서니科(Rubiaceae family) (Euasterid I clade of dicotyledonous plants)에 속하며, - 꼭두서니科는
⇒ 660 genera (屬),
⇒ 11,000 species (種) 이상으로 이루어져 있다 (Robbrecht & Manen, 2006). - 다소 젊은 科인 꼭두서니科(Rubiaceae)는 [90.4 My divergence time (Bremer and Eriksson, 2009)]
전세계적으로 분포한다;
그러나 가장 큰 종 다양성은 ⇒ (아)열대 지방((sub)tropics)에 집중되어 있다. - 그것은
⇒ 잎자루 사이에 턱잎(interpetiolar stipules)이 있는
단순한 對生葉(opposite leaves)으로 알아볼 수 있는,
⇒ 육생하는(terrestrial) 나무(trees), 관목(shrubs),
리아나(lianas, 열대산 칡의 일종), 또는 허브(herbs)로 구성된다. - Coffeeae 族(tribe)는
⇒ Ixoroideae (익소로이데아이, 치자나무 亞科) 단계통 아과(monophyletic subfamily)의 일부이며,
⇒ Gardenieae 및 Pavetteae 族(tribes)와 가깝다 (Bremer and Jansen, 1991). - Coffeeae tribe는
⇒ 전형적인 커피콩 형태, 즉 씨앗의 평평한 면에 홈이 있는 것이 특징이다. - 처음에는
┌ Coffea L.과
└ Psilanthus Hook f. 두 속(genera)이 ⇒ 개화 기준과 꽃 기준에 따라 구별되었다 (Bridson, 1982). - 각 속은
┌ 성장 습관(monopodial vs. sympodial (單莖性 vs. 假軸性) development)과
└ 꽃차례(inflorescence)의 유형(axillary vs. terminal flowers (겨드랑이 꽃 대 말단 꽃))을 기준으로
⇒ 2개의 亞屬 (subgenera)으로 나누어졌다. - 그러나 최근 연구들에 따르면
Coffea 種과 Psilanthus 種 사이의 서열 분화(sequence divergence) 수준은 무시할 수 있으며,
Coffea와 Psilanthus 種 사이에는 단 하나의 중요한 차이가 있음이 지적되었다;
Coffea는 ⇒ 길게 나와 있는 암술대(long-emergent style)를 가졌고,
Psilanthus는 ⇒ 짧고, 꽃 부리 밖으로 튀어나오지 않은 암술대(short, included style)를 가졌다. - 이러한 새로운 데이터를 기반으로 Psilanthus는
⇒ 최근 현재 Coffea 속에 포함되었다 (Davis et al., 2011). - 커피 종의 수는
⇒ 18세기 당시 알려진 유일한 종인 C. arabica로 Coffea L. 속이 생성된 이후 상당히 증가했다. - 19세기 말에는 ⇒ 약 15종이 기술되었으나,
다음 세기에 ⇒ 100종 이상의 새로운 종이 추가되었다.
지금까지 ⇒ 총 124종 (Coffea 104종, Psilanthus sp. 20종)이 기재되었다 (Maurin et al., 2007). - 새로운 종이 아직 기술되고 있기 때문에 커피 종 목록은 아직 완성되지 않았다.
- 더욱이, 커피 분류학에서 고려되는 많은 특성은 약하고 가변적이며,
많은 종의 특성이 완전히 규명되지 않았기 때문에, 이들의 관계에 대해 유효한 결론을 도출하기가 어렵다. - 커피나무 종은
⇒ 열대 및 아열대 지역에서 자연적으로 발생한다 (Fig. 1). - 예전 Coffea genus(屬)은
⇒ 기니(Guinea)에서 탄자니아(Tanzania),
⇒ 에티오피아(Ethiopia)에서 모잠비크(Mozambique)에 이르는 아프리카에 주로 존재한다.
⇒ 마다가스카르(Madagascar), 코모로(Comoros),
마스카렌 제도(Mascarene Islands) (Mauritius, Réunion)에도 존재한다. - 이전의 Psilanthus 屬은
⇒ 서부 아프리카에서 동부 아프리카까지(7種) 발견될 수 있지만 (Bridson, 1987; Davis et al., 2006),
⇒ 인도 (6種),
⇒ 동남아시아 (2種),
⇒ 오세아니아 및 북부 오스트레일리아 (5種)에서도 발견될 수 있다. - 3곳의 종 다양성 핫 스폿이
⇒ Madagascar (주로 동부지역의 상록 다습림),
⇒ Cameroon (Mount Cameroon) 그리고
⇒ Tanzania (주로 the Eastern Arc Mountains)에서 식별되었다 (Davis et al., 2006). - 분포 지역은 종마다 크기가 다양하다.
⇒ 아프리카에서는 서부 및 중앙 아프리카의 C. canephora 및 C. liberica와
⇒ 동아프리카의 C. eugenioides를 제외하고
⇒ 커피 種은 분포가 다소 제한되어 있으며, 때로는 몇 평방 킬로미터에 불과하다. - Madagascar에서는
⇒ C. perrieri만이 널리 분포되어 있다.
⇒ 59종의 마다가스카르 種 중 30종의 분포 규모는 지방(a province)과 유사하지만,
⇒ 나머지 28종의 경우 이 에리어는 숲이나 산으로 제한될 수 있다 (Noirot et al., 2016).

2.2 Habitat and adaptation
- 모든 커피 종은
⇒ 다년생 목본 灌木 또는 喬木(perennial woody bushes or trees)이며,
⇒ 형태, 크기 및 생태적 적응이 크게 다르다 (Fig. 2). - 커피나무는 다양한 유형의 열대우림(rainforest)에 서식해오고 있다 (Davis et al., 2006).
- 아프리카에서는, C. canephora, C. eugenioides, 그리고 C. liberica와 같이 널리 분포된 종은
⇒ 습한 상록수림 (humid, evergreen forest),
⇒ 갤러리 숲 (gallery forest, 강을 따라 띠 모양으로 조성된 숲),
⇒ 계절에 따라 건조한 상록수림 또는 낙엽-상록 혼합림(mixed deciduous-evergreen forest)에서 흔히 발견된다. - 또한,
⇒ 습한 상록수림(humid evergreen forest),
계류연안(rheophytic) (특히 sand banks 모래톱) 또는
계절/일시적으로 범람하는 하안림(riparian forest)의 C. congensis,
⇒ 운무림(cloud forest)의 C. fadenii,
⇒ 안정된 사구 위 연안림(littoral forest)의 C. racemosa (Davis et al., 2006), 그리고
⇒ 모래언덕과 산호초 기질(coral reef substrate)의 연안림(littoral forest)에 있는
C. pseudozanguebariae (Anthony et al., 1987) 등과 같이 특정한 적응들을 보여주기도 한다. - Madagascar에서는,
⇒ 종의 67%가 습한 상록수림(humid evergreen forests)에서만 자라는 반면,
⇒ 계절에 따라 건조한 숲에는 17%의 종이 포함되어 있다. - 다른 종들은 ⇒ 두 유형의 숲 모두에서 자란다 (Noirot et al., 2016).

- 숲에서의 종의 존재 여부는
계절별 및 연간 강수량 강도 뿐만 아니라,
최저 기온, 즉 대부분의 경우 고도와 위도에 따라 결정된다. - Madagascar에서는 커피나무가
⇒ 2,500m above sea level (asl)까지 존재하지만,
⇒ 그 범위 전체에 거쳐 존재하는 종은 없다. - 종의 27%는 ⇒ 해발 0~200m 사이에서 자라며,
61%는 ⇒ 해발 200~1,000m 사이에서 자란다. - 단 3 종만이 해발 50~1200m의 넓은 고도 분포를 가지고 있다 :
┌ C. dubardii,
├ C. hommlei, 그리고
└ C. perrieri (Noirot et al., 2016). - 아프리카에서도 비슷한 고도 범위가 관찰되었지만 (Fig. 3; Anthony et al., 2010),
해발 1000m 이상 자라는 종의 평균 비율은 마다가스카르보다 높다. - 커피 종들은
해발 최대 2300m (C. mufindiensis)에서 발견되지만,
대부분의 종(67%)은 1000m 미만의 제한된 고도 범위에 적응한다. - 6종은
┌ 저지대 (500m 미만)부터 1500m (C. brevipes, C. canephora),
├ 1800m (C. liberica, C. salvatrix),
├ 2000m (C. mongensis), 그리고
└ 2200m (C. eugenioides) 까지 넓은 고도 분포를 나타낸다.

2.3 Diversity of cultivated species
- 상업용 생두 생산은 C. arabica, C. canephora 및 C. liberica의 세 가지 주요 커피 종에만 의존한다.
- C. arabica (Arabica)와
C. canephora (Robusta)는 각각 전 세계 커피 생산량의 약 65%와 35%를 차지하지만,
C. liberica는 극히 일부만 재배된다. - C. canephora는 열대 저지대(0~800m asl)에 적응하는 반면,
C. arabica 종은 일반적으로 열대 고지대(600~1500m asl)에 적응한다. - 아라비카 커피는
로부스타 커피보다 더 온화하고 향이 더 강하며 카페인 함량이 적다. - C. arabica 종은
매우 낮은 유전적 다양성을 나타내지만 (분자 마커를 통해 추정한 바와 같이)
현저한 표현형 변이(phenotypic variation)를 나타내며,
특정 액세션들이 교배 되면 잡종 강세(hybrid vigour)가 강하다는
강력한 증거가 있다 (Van der Vossen et al., 2015). - 따라서
┌ 적응 능력,
├ 식물 높이 및 모양,
├ 잎 크기 및 모양,
├ 절간 길이,
├ 열매 색상 또는 모양,
├ 질병 저항성, 그리고
└ 소출 능력에서의 차이들과 같은 C. arabica 품종 간에 변이들이 관찰될 수 있다. - 이러한 다양성은 당연히 상당한 경제적 중요성의 요인들을 나타낸다.
- 대조적으로, C. canephora는
유전적으로 매우 다양한 종이다. - 자연적으로 서부 및 중앙아프리카의 습한 저지대에 널리 분포한다.
- 이 요인은 종의 적응 능력과 다양화의 차이에 유리했다.
- 엄격한 타화수분 성질(allogamous nature) (outcrossing만 발생)로 인해,
각 나무는 독특한 유전자형으로 간주될 수 있다. - C. liberica는
아프리카(Guinea)와 동남아시아(말레이시아, 필리핀, 베트남)의 특정 지역에서만 재배된다. - 이 종은 매우 다양하며,
┌ Dewevrei,
├ Excelsa,
├ Liberica 그리고
└ Dybowskii와 같은 많은 변종들을 포함한다. - 이러한 식물학적 변종들 중 일부는 가뭄에 매우 강하다(highly drought resistant).
- 나무의 크기(5~10m)가 걸출하여 재배를 어렵게 하며,
대량의 펄프가 가공 문제를 야기한다;
게다가 컵 품질은 일반적으로 로부스타 보다 열등한 것으로 여겨진다.
2.4 Molecular phylogenetics
- 분자계통발생학(Molecular phylogenetics)은
유기체의 진화 관계에 대한 정보를 얻기 위해,
주로 DNA 서열의 유전적인 분자적 차이(hereditary molecular differences)를 분석하는 계통발생학의 한 분야이다. - 지난 수십 년 동안 커피종의 몇 가지 분자 계통발생이
색소체(plastid) DNA의 유전자간 스페이서 서열(intergenic spacer sequences)의 변이
(Cros et al., 1998; Anthony et al., 2010),
rDNA의 ITS 서열(Lashermes et al., 1997),
색소체 영역(plastid regions)과 ITS의 조합 (Maurin et al., 2007; Davis et al., 2011), 그리고
색소체 영역과 핵 유전자(nuclear genes)의 조합(Nowak et al., 2012)에 기반하여 정립되어왔다. - 종 수준에서는,
분자 연구들에서 소수의 간결한 정보 특성(parsimony-informative characters)이 발견되었으며,
주요 계통군(primary clades)은 그 트리들에서 약하게 지지되었다. - 이는 Coffea 속의 최근 기원(few Mya)과
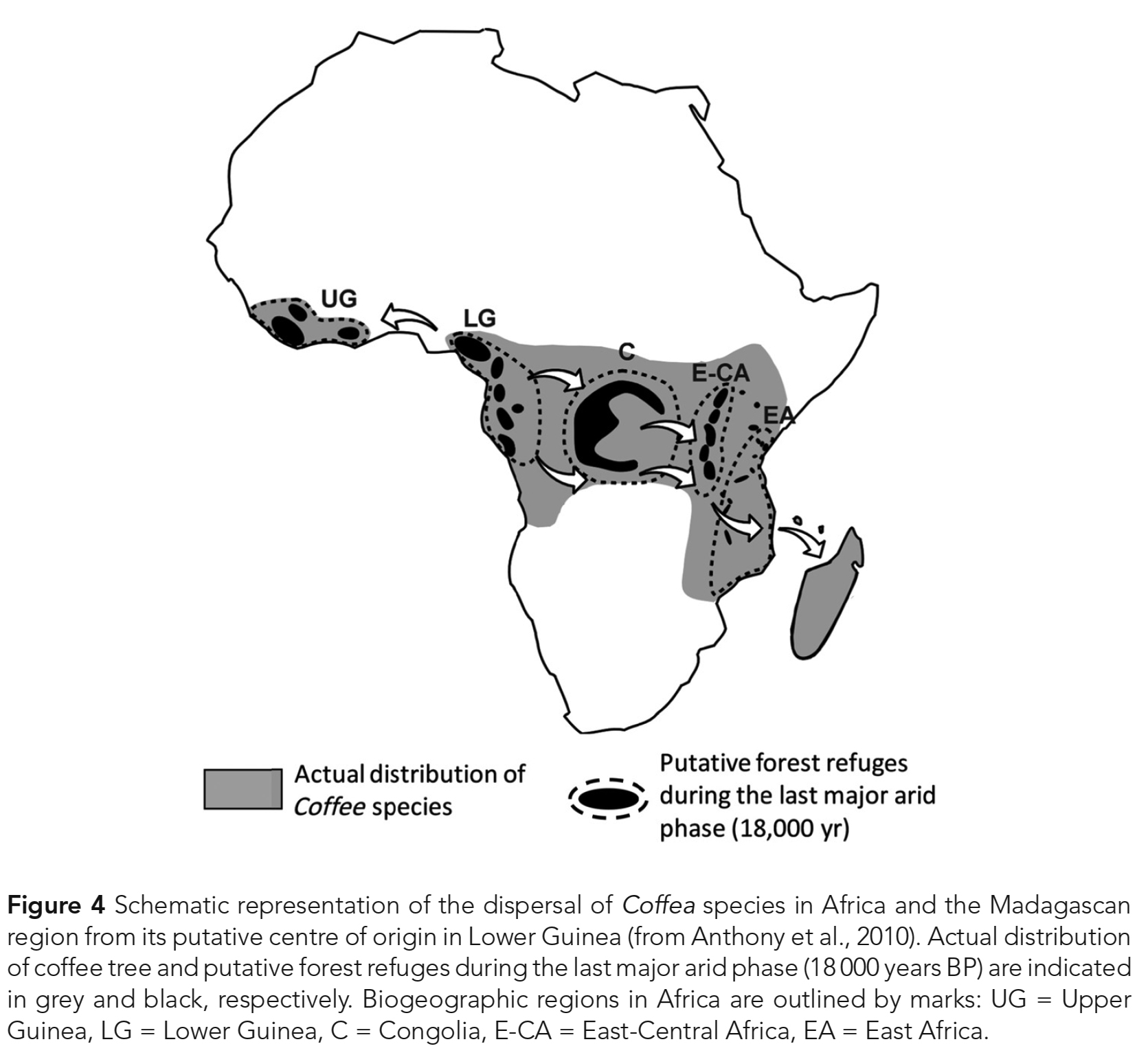
방사형 모드의 종 형성(radial mode of speciation)에 기인한다 (Anthony et al., 2010). - 모든 연구들이 종의 주요 그룹들과 그들의 지리적 기원 사이의 상응을 가리켰다.
- 지리적으로 일관된 여러 아프리카, 인도양 및 인도 계통군 (clades)이 잘 서포트되는 것으로 나타났다.
- 더욱이, 데이터는 서아프리카에 있는 Coffea의 원산지에 대한 생각과 일치했다
(Fig. 4; Anthony et al., 2010; Nowak et al., 2012). - 현재 Coffea의 일부이고 형태학적으로 다양한 종을 포함하는 아프리카 Psilanthus 그룹은
그 屬 내에서 초기 분기(early divergence)를 나타내는 것으로 제시되었다 (Nowak et al., 2012). - Madagascar의 대량서식은 아직 완전히 해결되지 않았다.
- 마다가스카르 종 간의 낮은 분기(low divergence)는
마다가스카르의 대량서식이 의심할 바 없이
아프리카 본토에서 발생한 몇 차례의 분산 사건(dispersal events)과
그에 따른 섬 종 형성(insular speciation)의 결과임을 가리킨다 (Anthony et al., 2010). - Nowaket al. (2012)은
마다가스카르가 아프리카 본토로부터의 두 가지 별개의 분산 사건(dispersal events)에 의해 대량서식화되었다는 생물지리학적 시나리오를 제안했다. - 인도양 코페아(Coffea) 종의 측계통성(paraphyly)에 대한 최초의 증거도 보고되었다.
- 특히, Mascarene 종은 잘 지지되는 단일계통군(monophyletic clade)을 형성하는 것으로 나타났다.
- 최근 81개의 Coffea 샘플 세트를 포함하는 대규모 계통발생 분석이 보고되었으며 (Hamon et al., 2017),
이는 이전 연구 결과를 확인하고 아프리카, 아시아, 마다가스카르 및 모리셔스 전역에 걸쳐 커피 속의 독립적인 전파(radiations)를 뒷받침한다.
3. Reproduction biology
- 식물이 외부교배(outcross) 꽃가루 이동을 위해 수분 매개자(pollinators)에 의존하는 정도는
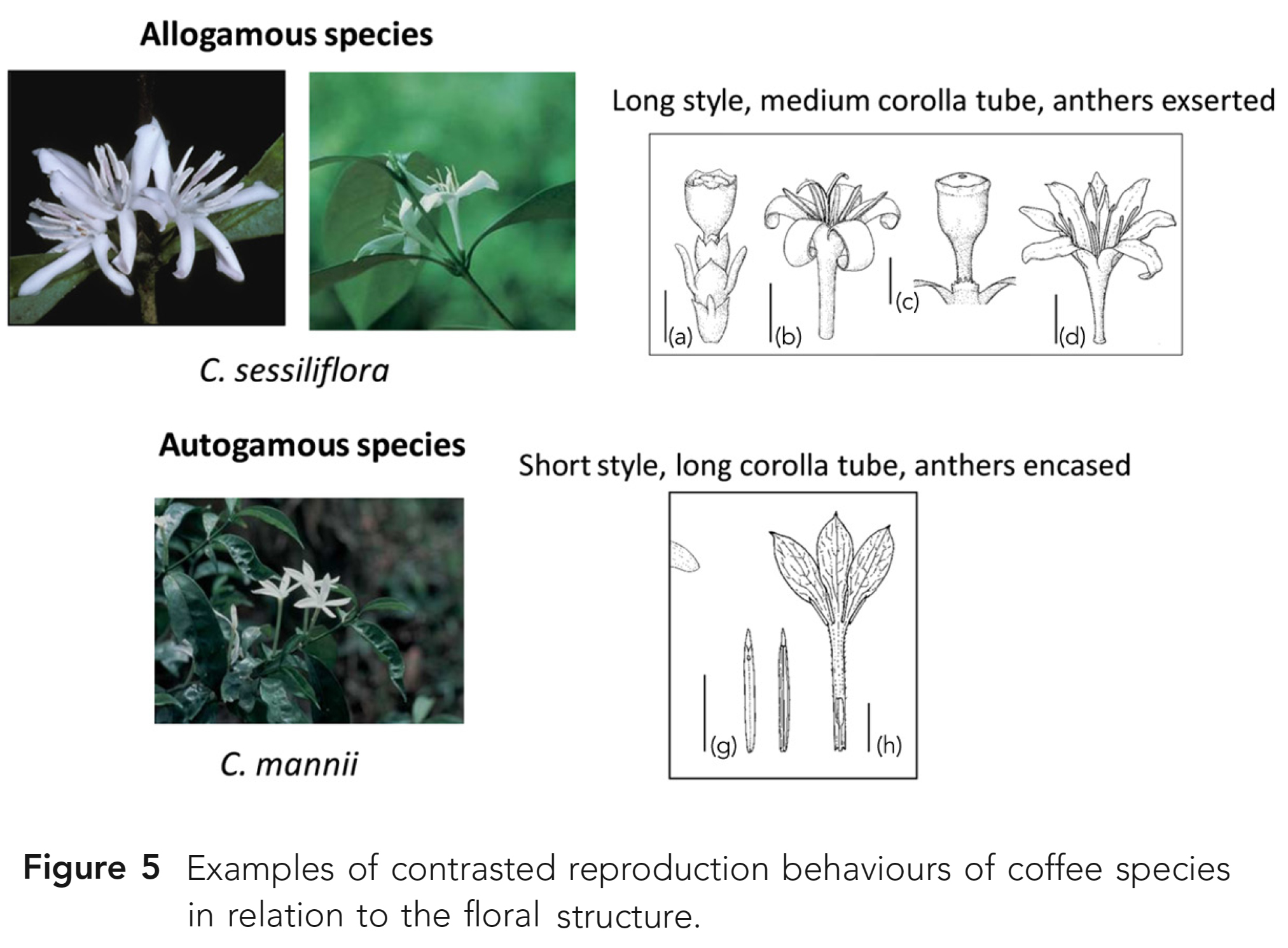
식물 생식 과정에서 중요한 문제이다. - 커피종과 관련하여 대조되는 번식 행태들이 보고되어왔다 (Fig. 5).
自家不和合性 & 自家和合性
- 커피 종은 일반적으로
배우체 시스템의 불화합성(gametophytic system of incompatibility)과 관련하여
自家不和合性(self-incompatible)이다 (Berthaud, 1980; Lashermes et al., 1996; Nowak et al., 2011). - 커피 자가불화합성(self-incompatibility (SI)) 메커니즘은
비-자기 꽃가루 관(non-self pollen tubes)이 암술대(style)를 통해 성장할 때,
RNase 세포독성(cytotoxicity)의 선택적 비활성화(selective deactivation)를 통해 기능하는
S-RNase 시스템에 의존한다 (Nowak et al., 2011). - S-RNase GSI 시스템은
생화학적으로 특성화된 최초의 SI 메커니즘 중 하나였으며,
asterid (菊花群) (Solanaceae (가지科), Plantaginaceae (질경이科)) 및 rosid (장미군) (Rosaceae (장미科)) 계통
모두의 기능적 특성에 의해 입증된 바와 같이,
조상 쌍떡잎식물(ancestral eudicot) 조건을 나타낼 가능성이 높다. - 그러나 이전 Coffea 屬에 속하는 대부분의 커피 종들은
자가-불화합성(self-incompatible)이지만,
자가-화합성(self-compatibility)을 가져온 주목할만한 예외가 있다는 점에 주목해야 한다. - 자가-화합성(self-compatibility)은
C. heterocalyx (Coulibaly et al., 2002)와 C. anthonyi (Stoffelen et al., 2009)라는
두 가지 이배체 (diploid) 종에서 확인되었다. - 마찬가지로, C. arabica는 자가-화합적(self-compatible)이다.
- 다른 속씨식물 계통(angiosperm lineages)에서
이전에 기록된 배수성(polyploidy)과 SI 분해(breakdown of SI) 사이의 강한 상관관계를 감안하면,
C. arabica의 자가-화합성(Self-compatibility)은 놀라운 일이 아니다. - 결과적으로, C. arabica는
C. arabica 재배품종들에서 약 10%의 타가교배율(outcrossing rate)을 보이는
주로 자가생식 종(selfing species)이라고 널리 인식되고 있다 (Van der Vossen et al., 2015). - 그러나 최근 야생 아라비카 커피 개체군들에서 혼합교배종(mixed mating species)의
전형적인 타가교배율(outcrossing rate)이 76%에 달하는 것으로 보고되었다 (Berecha et al., 2014). - 더욱이, 自花受粉 (autogamy)은
이전 Psilanthus 亞屬(subgenus)의 모든 종들에도 적용된다. - 시험된 유일한 종인 C. bengalensis는 自家受精(self-fertile)이 가능하다 (Charrier pers. comm.).
- 또한, 해당 종은 부분적으로 꽃밥이 포함된(partly included anthers) 짧은 암술대(short stylus)가 있는
꽃 구조를 보여주며, 이는 절대 자가수정 종(obligate selfing species)의 전형적인 특징이다.
受粉
- 커피 나무에는 바람과 벌의 수분(wind and bee pollinations)이 공존한다 (Noirot et al., 2016).
- 예를 들어, C. canephora의 경우
화창한 날에는 풍매 수분과 충매 수분(anemophilous and entomophilous)이 모두 이루어지며,
비가 오는 날에는 풍매 수분이 된다. - 실제로 바람과 벌 수분 사이의 이러한 대조는 종마다 다르다:
예를 들어 C. pseudozanguebariae와 C. mauritiana에서는 바람 수분을 선호하는 꽃이 나무에 분산되어 있다. - 이와 반대로 C. canephora와 C. arabica에서는 꽃이 무리를 지어 벌의 매력을 높인다.
- 모든 중간 상황들이 존재하며, 환경에 대한 중요한 특정 적응을 나타낸다 (Noirot et al., 2016).
- 서로 다른 커피 종 간의 교차수분(Cross-pollination)과 잡종 생산이 가능하다.
- 자연 조건에서는, 자발적인 잡종의 발생이 거의 관찰되지 않았다.
- 다른 접합 전 장벽(pre-zygotic barriers) 중에서 개화 중첩(flowering overlap)이 없으면
일반적으로 종간 교잡화(interspecific hybridization)를 방지하는 것으로 생각된다. - 그러나 여러 종이 함께 재배되면 잡종이 발생할 수 있다
- 예를 들어 Timor Island (Goncalves and Rodrigues, 1976)과
New Caledonia (Mahe et al., 2007)에서는 Timor Hybrid. - Coffea 종 간의 실험적 교잡화는 널리 보고되었으며,
비교적 번식력이 좋은 하이브리드들을 생산했는데,
이는 완전한 접합 후 분리 장벽(post-zygotic isolation barriers )이 부족함을 나타낸다. - 특정 이종간 F1 하이브리드들의 생식력 감소가
부모 종의 핵 함량(nuclear content)의 상당한 차이와 연관되어 있는 것으로
나타났음에도 불구하고 (Barre et al., 1998),
전반적인 데이터는 커피 종 간의 분기(divergence)가 낮고,
게놈이 강한 친화력(strong affinities)을 나타내고
염색체 재배열(chromosomal rearrangements)이 제한되어 있음을 나타낸다.
4. Genome organization and evolution
- 커피 종의 기본적인 염색체 수는 x = 11 chromosomes인 것으로 여겨지며,
이는 Rubiaceae 과의 대부분의 속들에서 전형적이다 (Kiehn, 1995). - C. arabica를 제외하고(allotetraploid with 2n = 4x = 44 chromosomes),
모든 Coffea 속의 종들은 이배체이다 (2n = 2x = 22 chromosomes). - 커피의 체세포의 유사분열 중기 염색체들(Somatic metaphase chromosomes)은 비교적 작으며 (1–3 μm)
서로 형태적으로 비슷하여 (Krug and Mendes, 1940; Bouharmont, 1959)
종에서의 거의 차이를 보이지 않는 균일한 핵형(karyotypes)을 초래한다. - 감수분열의 파키틴期 단계(meiotic pachytene phase)에서의 관찰로
상당히 더 나은 염색체 특성 분석을 할 수 있었다(Pinto-Maglio and Cruz, 1998). - 커피 염색체들을 세로 방향으로 구별하려는 최초의 성공적인 시도는
파키텐 염색체 패턴(pachytene chromomeric patterns)의 매핑에 의해서, 그리고
밴딩(banding) 및 FISH 테크닉에 의해서 이루어졌다 (Pinto-Maglio, 2006). - C. canephora 게놈(710Mb)의 최근 시퀀싱은
커피 게놈 구조와 조직 뿐만 아니라
이 주요 작물의 생물학과 진화의 중요한 측면을 탐구할 수 있는 명확한 수단을 제공한다 (Denoeud et al., 2014). - 전이 가능한 요소(transposable elements, TE)의 컴퓨터 식별이 수행되었다.
- TE는
게놈 내에서 포지션을 바꿀 수 있는 DNA sequence로,
때로는 돌연변이를 생성하거나 역전시키고 세포의 게놈 크기를 변경한다. - TE는
이용가능한 커피 게놈 서열의 절반 이상을 나타내며,
그중 LTR-RT는 가장 빈번한 요소들의 순서이다 (게놈의 42%). - 몇몇 커피 종의 nuclear DNA 함량이 유세포 분석법(flow cytometry)을 통해 추정되었다.
- 다른 속씨식물(angiosperms )과 비교할 때 (Bennett and Leich, 1995),
커피 종의 게놈 크기는 다소 작은 것으로 보인다. - 그러나 Coffea 종에서는 이배체 게놈 크기(diploid genome sizes)의 주목할만한 변이가 관찰된다.
- 이 변이 범위는 469에서 900Mb이며.
아프리카의 동쪽에서 서쪽으로 (Noirot et al.,2003), 그리고
마다가스카르에서는 북쪽에서 남동쪽으로 (Razafinarivo et al., 2012) 게놈 크기가 증가하는 일반적인 패턴이며,
이는 그 종의 종분화(speciation) 및 적응 과정 하에서 핵 DNA가 점진적으로 축적되었음을 시사한다. - 식물 게놈 크기가 Tes의 비율과 직접적으로 연관되어 있다는 것은 잘 알려져 있다.
- 커피에서는, 예비 분석에 따르면
소수의 TE families의 빠른 확산(proliferation)이 이러한 게놈 크기 차이를 설명하는데 관여되지 않는다는 것을 보여주었다. - 그러므로 그 게놈 크기 변화(genome size variation)는
대규모 패널의 families에 속하는 수많은 TEs (주로 LTR-RT)의 차별적 축적으로 인해
발생한 것일 수 있다고 제시되었다 (Guyot et al., 2016). - 커피 게놈에는 상대적으로 적은 수의 유전자가 포함되어 있다.
- C. canephora에서는
약 25,574개의 단백질-코딩 유전자들이 식별되었으며,
따라서 이질사배체(allotetraploid) 아라비카 게놈에서는 약 50,000개의 유전자가 있을 것으로 예상할 수 있다. - 그럼에도 불구하고, 잠재적인 적응 가치를 지닌 커피 특유의 유전자군 확장이 보고되었다.
- 이러한 확장된 계열에는
카페인 생산에 관여하는 N-methyltransferases (NMTs), 방어 관련 유전자, 그리고
2차 화합물 합성에 관여하는 알칼로이드 및 플라보노이드 효소가 포함된다는 점은 주목할 만하다. - 카페인 NMTs의 비교 분석은
이러한 유전자들이 카카오 및 차의 유전자들과는 독립적으로 순차적인 직렬 복제(sequential tandem duplications)를 통해 확장되었음을 보여주며, 이는 진정쌍떡잎식물(eudicots)의 카페인이 다계통 기원(polyphyletic origin)임을 시사한다 (Denoeud et al., 2014). - 구조적으로 커피 게놈은
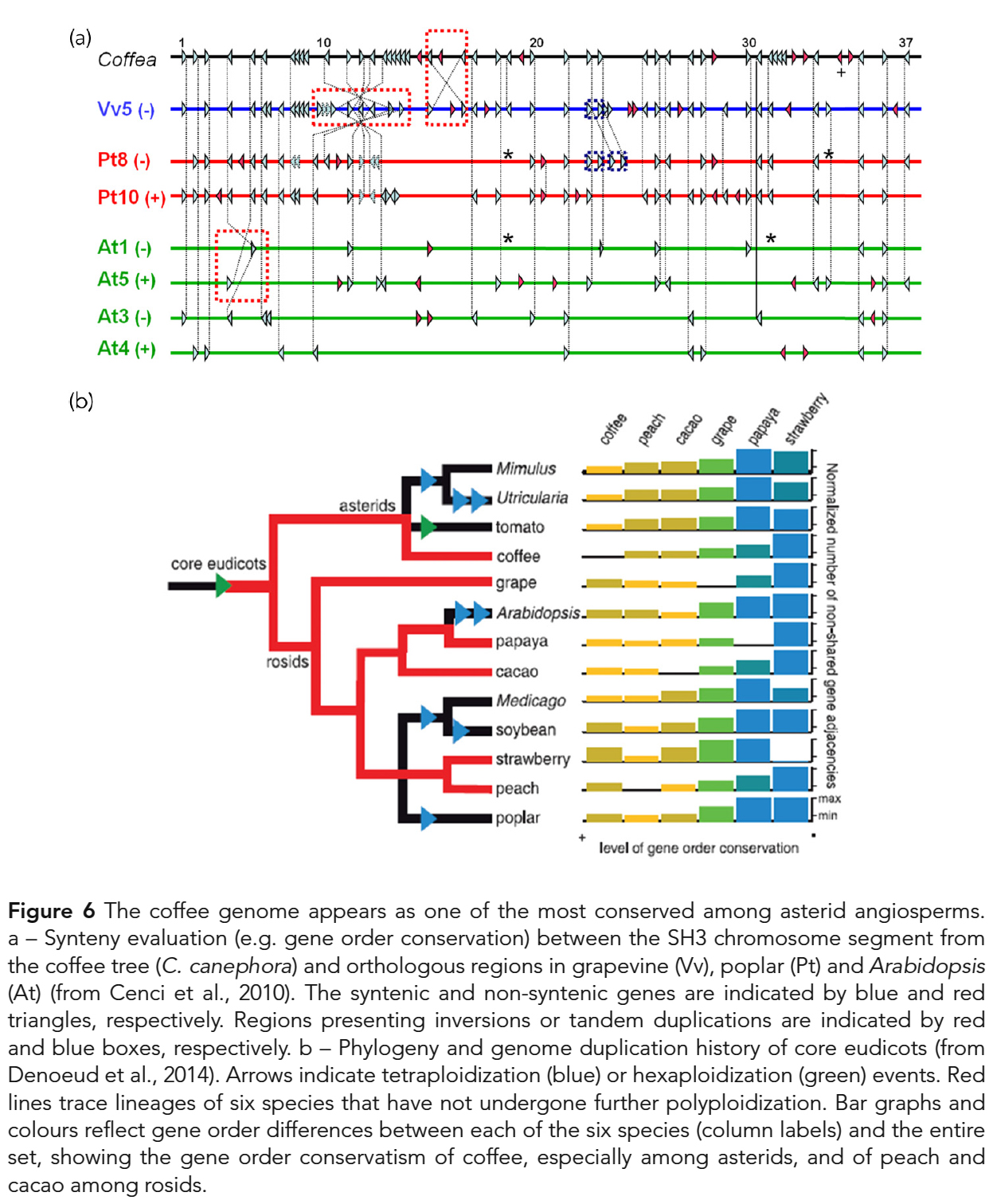
국화군 속씨식물(asterid angiosperms) 중에서 가장 보수적인 염색체 유전자 순서(chromosomal gene order)를
나타낸다는 점을 고려할 때 안정적이고 보수적인 것으로 보인다 (Fig. 6; Denoeud et al., 2014). - 더욱이, 약 1억 1천만년전 핵심 진정 쌍떡잎 식물(core eudicots)의 기원에서
γ 삼중증식(triplication)이 이루어졌기 때문에,
커피 게놈은 그 계보에서 전체 게놈 중복(whole-genome duplication)의 징후를 보이지 않는다 (Cenci et al., 2010). - 따라서 커피 게놈은
토마토와 감자와 같은 Solanum (가지屬) 종에서 설명된 전체 게놈 삼중화(whole-genome triplication)를
보여주지 않으며,
이는 커피 나무와 Solanum 사이의 마지막 공통 조상이 이 사건보다 앞서 있음을 나타낸다. - 또한, 異種相同性遺傳子 (orthologous genes)에 의해 코딩되는 단백질의 서열 분석(sequence analysis)을 통해
Coffea 단백질의 진화 속도(evolution rate)를 다른 종과 비교하였다 (Cenci et al., 2013). - 국화군 계통군(asterid clade)에 속하는 종 중에서, C. canephora는
다른 종(Solanum lycopersicum 및 Mimulus Guttatus)보다 더 느리게 진화한 것으로 보인다. - 이러한 발견은
다년생 습관(perennial habit)과 느린 진화 속도(slow evolution rates) 사이의 상관 관계와 일치적이다.
5. Arabica origin and diversification
- C. arabica는 에티오피아 남서부의 고지대와 수단의 보마 고원에 유전적 다양성의 주요 중심지가 있다.
- C. arabica의 야생 개체군은
Mount Imatong (수단)과
Mount Marsabit (케냐)에서도 보고되었다 (Thomas, 1942; Anthony et al., 1987). - C. arabica의 재배는 약 1500년 전에 에티오피아 남서부에서 시작되었다.
- 현대의 커피 재배품종들은
18세기에 전 세계적으로 퍼진 C. arabica의 Typica와 Bourbon으로 알려진 두 가지 기본 개체군에서 파생되었다 (Anthony et al., 2002). - 역사적 데이터에 따르면 이러한 개체군은
매우 소수의 개체들, 즉 Typica 개체군의 경우 단 한 개체만 존재하고,
부르봉 개체군의 경우에는 1715년과 1718년에 the Bourbon Island (현재 레위니옹)에 도입된
소수의 나무들로 구성되었다.
5.1 An allotetraploid genome
- C. arabica의
게놈 구성(genome constitution)과 種分化 方式(mode of speciation)은 여러 차례 조사되었다. - Restriction fragment length polymorphism (RFLP) 유전자좌 마커(loci markers)와,
Genomic in situ hybridization (GISH)를 조합 사용하여
C. arabica의 기원을 조사했다 (Lashermes et al., 1999). - 잠재적 이배체 조상 종(potential diploid progenitor species)의 RFLP 패턴을
C. arabica의 RFLP 패턴과 비교함으로써,
C. arabica에서 결합된 두 세트의 염색체 또는 게놈의 원천(source)이 특정되었다. - C. arabica의 게놈 조직(genome organization)이
두 개의 추정 게놈 공여자 종들(putative genome donor species)로부터
동시에 표지된 전체 게놈 DNA(simultaneously labelled total genomic DNA)를 프로브(probes)로 사용하여
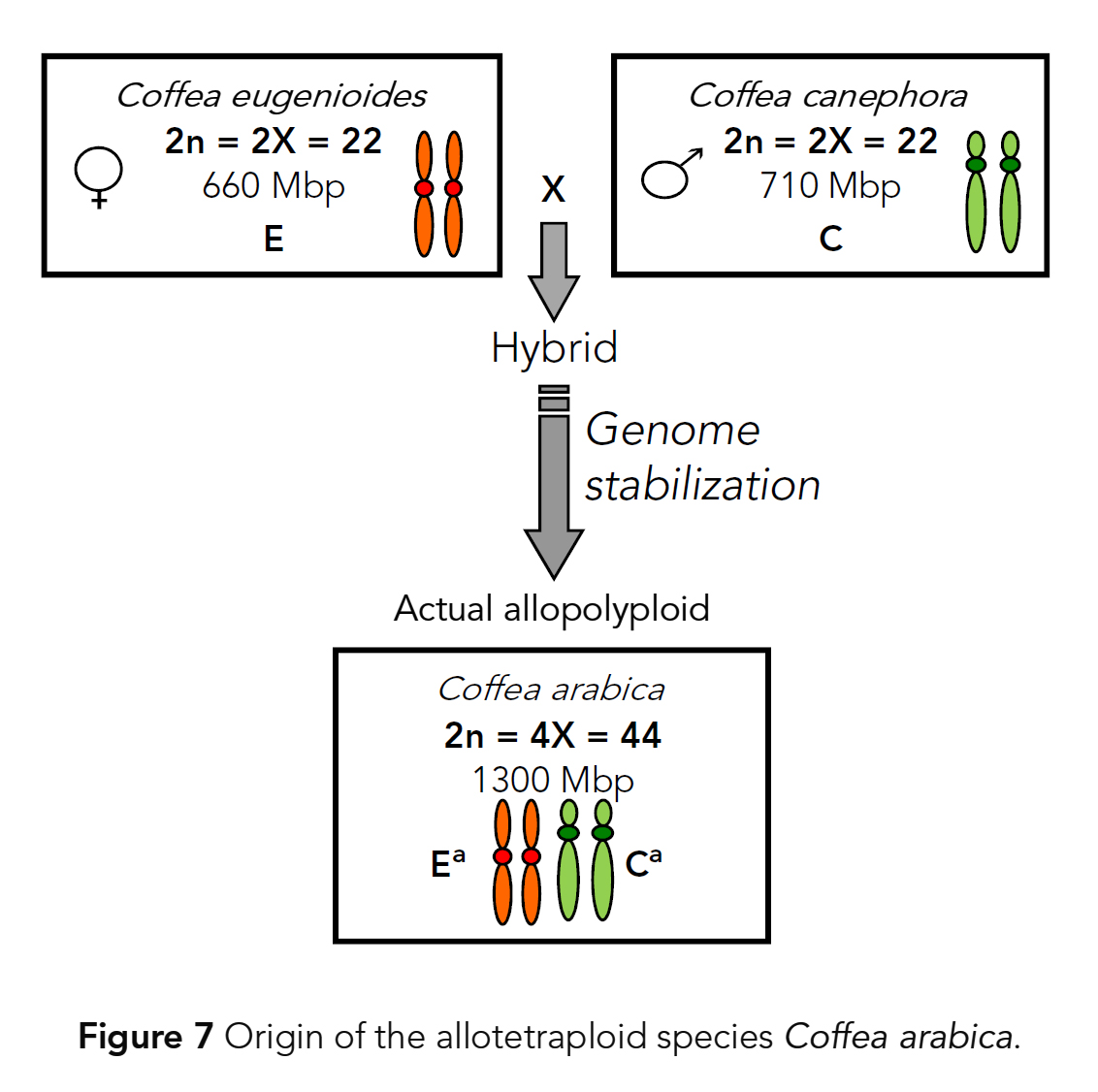
GISH에 의해 확인되었다 (Raina et al., 1998; Lashermes et al.,1999). - C. arabica는
두 개의 이배체 종인 C. canephora(genome C)와 C. eugenioides(genome E) 사이의
교잡화(hybridization)에 의해 형성된
이질사배체 게놈(allotetraploid genome)(genome CaEa)을 갖는 것으로
나타났다 (Fig. 7; Lashermes et al., 1999). - 커피에서 엄격한 모계 cpDNA 유전을 가정할 때 (Lashermes et al., 1996b),
색소체 변이 분석(plastid variation analysis)은
C. eugenioides가 C. arabica의 모계 조상 종이었음을 나타낸다 (Cros et al., 1998). - C. arabica의 두 이배체 구성 하위 게놈(diploid constitutive subgenomes)
(각각 C. canephora 및 C. eugenioides에서 파생된 두 세트의 상동 염색체)과
그것의 현대의 조상 종(progenitor species)의 게놈 사이의 낮은 분기(low divergence)는
종분화 과정(speciation process)이 매우 최근에 발생했음을 시사한다 (약 50,000년 전; Cenci et al., 2012).
- 커피 종의 현재 분포를 기반으로 C. arabica의 종분화 과정(speciation process)을
중앙 아프리카에서 정확하게 파악하는 것은 어려운 것으로 보인다. - 두 부모 종 모두 이종간 교잡화(interspecific hybridizations)가 발생했을 수 있는
중앙 아프리카 고원의 서쪽에서 동일하게 관찰되었다 (Thomas, 1944). - 그러나 중앙아프리카 열대우림의 구성과 범위는
(신생대) 제4기 후기(late quaternary period)의 환경 변화에 크게 영향을 받았기 때문에
과거에는 다른 접촉 지역을 배제할 수 없었다. - 더욱이,
두 개의 구성 게놈(two constitutive genomes) (동조 염색체의 두 세트) 사이의 밀접한 관계에도 불구하고,
감수분열(meiosis)에서는 二價(bivalents)가 관찰되며,
C. arabica는 이배체-같은 감수분열 행태(diploid-like meiotic behaviour)를
보이는 것으로 생각된다 (Krug and Mendes, 1940). - 조사 결과 C. arabica에서는
동조 염색체가 쌍을 이루지 않는 것으로 나타났는데,
이는 구조적 분화의 결과(structural differentiation)가 아니라
쌍 조절 인자(pairing regulating factors)의 작용 때문이다 (Lashermes et al., 2000).
5.2 A single origin of C. arabica
- 배수성 종(polyploidy species)의 형성에서 한 가지 중요한 측면은
독립적인 기원의 수(number of independent origins)이다. - 많은 이질배수체 종들(allopolyploid species)은
여러 가지 독립적인 교잡화 사건들(hybridization events)로 인해
여러 기원을 갖는 것으로 여겨진다 (Soltis et al., 2003; Doyle and Egan, 2010). - 그러나 C. arabica의 경우,
모든 C. arabica의 액세션들이 공유하는
다수의 동조의 교환 사건(homoeologous exchange events)에 대한 증거는
단일 이질배수체화 사건(single allopolyploidization event)에 대한 가설을
강력하게 뒷받침한다 (Lashermes et al., 2014, 2016). - 모든 C. arabica 개체군들은
크고 특정한 동조 교차 교환(large and specific homeologous crossover exchanges)과 관련된
독특한 이질배수체화 사건(allopolyploidization event)으로부터 파생된다.
5.3 Genome plasticity and diversification
- 여러 조사에서, 원래의 교잡화 사건(original hybridization event) 이후,
C. arabica의 전반적인 구조적 게놈 안정성 (overall structural genome stability)이 지적되었다. - 아라비카(C. arabica)에서
두 개의 완전한 동조 염색체 세트의 존재와 관련된
고정된 이형접합성(fixed heterozygosity) 상태가 보고되었다 (Lashermes et al., 2014). - 그러나
동조적 DNA 교환(homeologous DNA exchanges) 및
동조체 침묵(homeolog silencing) (두 동조체 중 하나의 검출 가능한 전사체가 없는 것으로 정의됨)이
조상 이질사배체(the ancestral allotetraploid)의 안정화 및 생존과 그리고
그것의 후속 다양화 (diversification)에 중요한 역할을 할 수 있다는 것이 입증되었다. - 진화의 초기 단계는 주로 동조성 교차 교환(homeologous crossover exchanges)을 포함했지만,
후기 단계에서는 유전자 전환(gene conversion) 및 동조체 침묵(homeolog silencing)을 포함하는
중복 유전자 진화(duplicate gene evolution)의 보다
점진적인 단계에 의존한 것으로 보인다 (Lashermes et al., 2016). - 이질배수체(allopolyploids)의 분자 데이터는
유전적 및 후생적 변화(genetic and epigenetic changes)가
다양한 종에 걸쳐 다배체화(polyploidization)의 일반적인 결과임을 나타낸다
(Madlung and Wendel, 2013; Song and Chen, 2015). - 그럼에도 불구하고 우리는 이러한 변화를 초래하는 메커니즘을 거의 이해하지 못하고 있으며,
그 변화의 방향성이나 무작위성에 대해서는 훨씬 더 적게 알고 있다. - 이들의 적응적 역할은 아직 확립되지 않았지만,
이러한 변화가 다양성의 주요 원천이며
C. arabica의 다양화에 중요한 역할을 했다는 것은 의심할 여지가 없다 (Fig. 8).
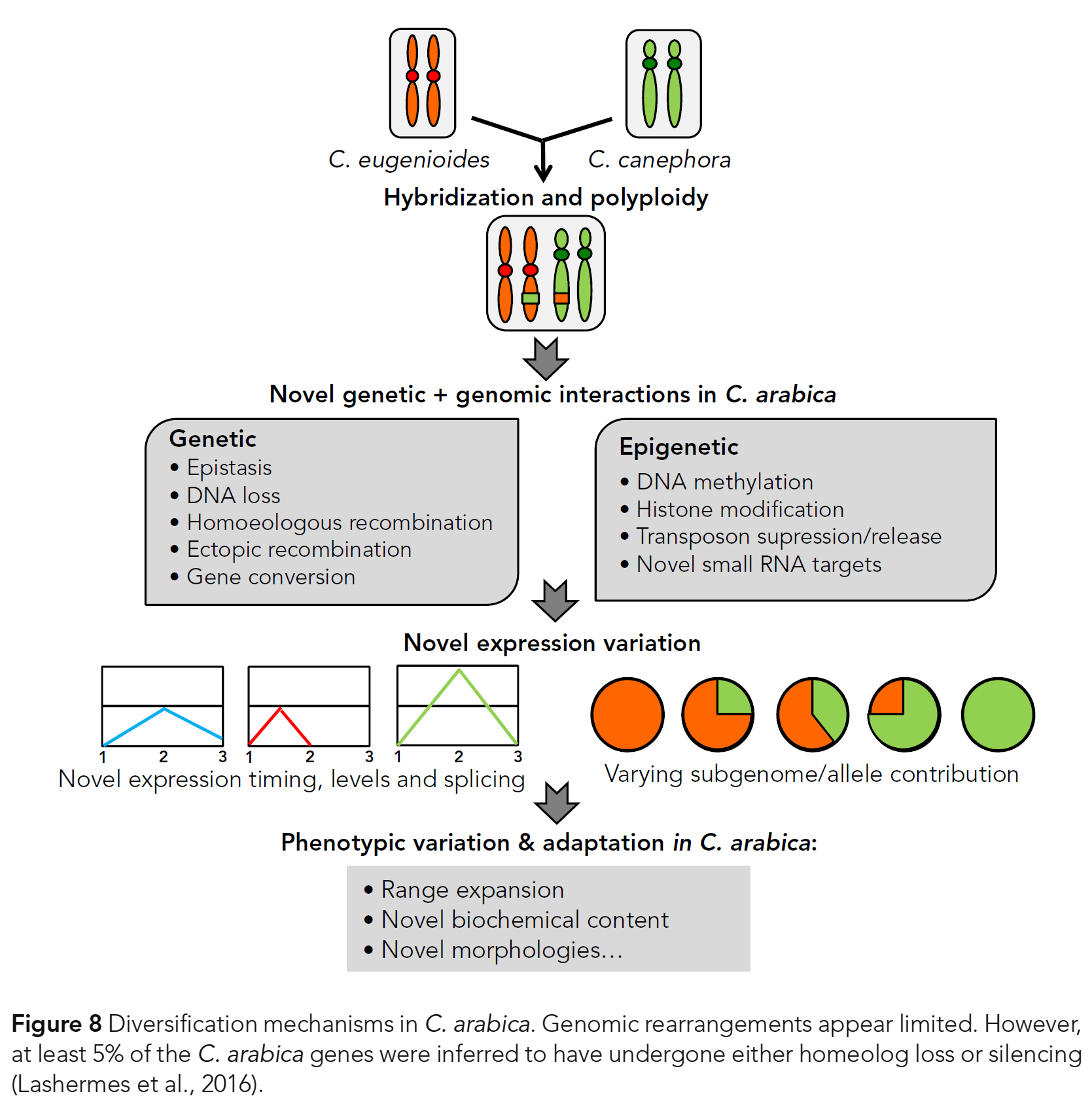
- 유전자 발현의 변화(Gene expressions’ changes)는 또한
이질배수체(allopolyploidy)의 일반적인 결과이며,
중복 유전자(duplicate genes)의 기능적 가소성은
배수체(polyploids)의 환경 적응에 중요한 역할을 하는 것으로 여겨진다 (Jackson and Chen, 2010; Song and Chen, 2015). - C. arabica에서 동조 발현(homeologous expression)의 변화가 조사되었다.
- C. arabica 및 관련 모종(parental species)의 게놈 전체 발현 패턴(genome-wide expression pattern)을 비교했다.
- 주어진 유전자에 대해 동조체의 전체 발현(total expression of homeologs)이
배수체 부모(polyploid parents) 중 단 하나와 통계적으로 동일한 경우에,
이질배수체 (allopolyploid)의 발현 조건을 설명하는
게놈발현 우성(Genomic expression dominance)이 관찰되었고,
성장조건들에 의해 조절되는 것으로 나타났다 (Bardil et al., 2011). - 그러나 두 하위 게놈(two subgenomes) 중 어느 것도 우선적으로 발현되지 않으며,
C. arabica의 전반적인 유전자 발현은
뒤얽힌 메커니즘(intertwined mechanisms)에 의해 조절되는 것으로 보인다 (Combes et al., 2013). - Combes et al. (2015)은,
종간 Coffea 하이브리드들을 사용하여,
유전자 발현 유전 패턴(gene expression inheritance patterns)과
특히 발현 수준 우세(expression level dominance)는
부모 대립유전자 간의 조절 차이(regulatory divergences)에 의해 결정된다고 보고했다. - 더욱이, 이질배수체(allopolyploid ) C. arabica의 더 나은 전사 항상성(better transcriptional homeostasis)은
이배체 부모 종에 적합하지 않은 환경에 직면했을 때
더 큰 표현형 항상성(greater phenotypic homeostasis)을 제공한다고 제시되었다 (Bertrand et al., 2015). - 유사하게, 비생물적 스트레스(abiotic stress)에 반응하여 동조체(homeologs)의 차등적 기여가 관찰되었다 (Marraccini et al., 2011; de Carvalho et al., 2014).
- 그러나 변화하는 환경에 반응하여
C. arabica의 기능적 가소성(functional plasticity)에 대한 이 메커니즘의 실제 기여는 깊이 조사되어야 한다.
6. Future trends and conclusion
기후 변화와 천연자원의 지속 불가능한 사용의 부정적인 영향의 관점에서, 커피 생산은 진화해야 할 것이다. 이는 특히 생산 수명이 매우 길고 급속한 유전적 획득이 지루한 커피와 같은 다년생 식물과 관련이 있다. 20세기에 커피 생산은 자연발생적인 아라비카 돌연변이(예: 소형 품종 Caturra), 종 간 천연 하이브리드들 (예: 티모르 잡종), 그리고 생산성이 높은 카네포라 클론의 선택을 통해 이익을 얻었다. 커피 생물다양성을 성공적으로 활용한 이러한 사례들은 상당한 영향을 미쳤지만 특히 이용 가능한 거대한 다양성과 관련하여 여전히 제한적이다.
유전적 다양성을 포착 및 조작하고 육종 프로그램에서 생식질을 효과적으로 사용하는 능력은 지속가능한 커피 생산에 필수적이다. 최근 게놈 분석을 위한 고용량 방법의 개발은 광범위한 의미를 지닌 새로운 패러다임을 제시한다. 재배되는 종 C. arabica와 C. canephora에 대한 대규모 분자 유전체학의 출현으로 이전에는 접근할 수 없었던 유전적 변이의 원천에 접근할 수 있게 되어 육종 프로그램에 활용될 수 있게 될 것이다.
예상되는 결과는 다음과 같다
(i) 유전자원의 신속한 특성분석과 관리,
(ii) 중요한 특성의 유전적 조절에 대한 이해 강화,
(iii) 관심 특성의 기초가 되는 후보 유전자 또는 밀접하게 연결된 게놈 영역의 식별, 그리고
(iv) 고려되는 특성에 유리한 영향을 미치는 게놈 영역의 변이체 또는 후보 유전자의 대립 유전자가 포함된 유전자 컬렉션의 액세션들의 식별.
그러기 위해서는 생물다양성에 기반한 발견에서 파생된 이익이 원산지 국가와 공평하게 공유되도록 보장하는 국제적 협력과 합의가 매우 적합하다.
야생 커피 개체군에 대한 우려는 세계에서 재배되는 대다수의 커피가 환경적으로 취약한 좁은 유전자 풀 (narrow genetic pool)을 가진다는 것을 상기할 때 나온다 (Mehrabi and Lashermes, 2017). 최근 몇 년 동안 우리는 중앙아메리카에서 커피잎녹병(Hemileia vastatrix)이 널리 퍼지는 것을 목격했다. 또한 지난 2년 동안 최근 가뭄으로 인해 브라질이 전 세계 생산량에서 차지하는 비중이 30%로 급락했다 (Bunn et al., 2015). 그럼에도 불구하고 유전자 자원 보전에 대한 관심은 아직까지 미흡한 실정이다.
야생 커피의 자연 서식지는 수십 년 동안 아프리카와 마다가스카르에서 발생한 삼림 벌채로 인해 위협을 받아왔다 (Andrianasolo et al., 2013; Gomez et al., 2009). 예를 들어, 에티오피아의 C. arabica 유전자 자원에 대한 조사에서는 부적절한 보전 노력과 삼림 벌채로 인해 유전자 풀의 유전자 침식 비율(rate of genetic erosion)이 놀랄 만큼 높다는 사실이 밝혀졌다 (Woldermariam et al., 2002). 야생 C. arabica 유전자형의 지리적 패턴화 및 분포는 에티오피아 야생 커피의 현장 보존을 위한 다중 현장 접근 방식이 필요함을 더 의미한다 (Tesfaye et al., 2014).
또한 야생 커피 유전자 자원 보존에 대한 인적 관리 및 토지 이용 결정의 부정적인 영향은 기후변화로 인해 더욱 악화된다.
21세기 동안 아라비카 커피 재배에 적합한 지역은 최대 40%, 최악의 경우 90%까지 감소할 것으로 추정된다 (Davis et al., 2012). 또한 야생 커피 숲에는 자연환경에서 해충과 질병이 얼마나 중요한지 통제되는 생태학적 세부 정보도 포함되어 있다.
따라서 이러한 야생 커피 숲은 현재까지 거의 개발되지 않은 자원인 지속가능한 해충방제 방법을 개발할 수 있는 방법에 대한 통찰력을 보유할 가능성이 높다. 또한 천연 야생 아라비카 커피 개체군을 보존하면 커피 유전자 자원의 현장 보존이 보장될 뿐만 아니라, 향후 저-투입 농업에 적용할 수 있어 높은 菌根 多樣性(high mycorrhizal diversity)을 보호할 수 있다 (De Beenhouwer et al., 2015).
7. Where to look for further information
Angiosperm phylogeny website: https://www.mobot.org/MOBOT/research/APweb/
Coffee genome resources: https://coffee-genome.org/
8. References
- Andrianasolo, D. N., Davis, A. P., Razafinarivo, N., Hamon, S., Rakotomalala, J. J., Sabatier, S. A. and Hamon, P (2013), High genetic diversity of in situ and ex situ populations of Madagascan coffee species: further implications for the management of coffee genetic resources, Tree Genet. Genomes, 9, 1295–312. doi:10.1007/s11295-013-0638-4.
- Anthony, F., Berthaud, J., Guillaumet, J. L. and Lourd, M. (1987), Collecting wild Coffea species in Kenya and Tanzania, Plant Genet. Resour. Newsl., 69, 23–9.
- Anthony, F., Combes, M. C., Astorga, C., Bertrand, B., Graziosi, G. and Lashermes, P. (2002), The origin of cultivated Coffea arabica L. varieties revealed by AFLP and SSR markers, Theor. Appl.Genet., 104, 894–900.
- Anthony, F., Diniz, L. E. C., Combes, M. C. and Lashermes, P. (2010), Adaptive radiation in Coffea subgenus Coffea L. (Rubiaceae) in Africa and Madagascar, Plant Syst. Evol., 285, 51–64. doi:10.1007/s00606-009-0255-8.
- Bardil, A., Dantas de Almeida, J., Combes, M. C., Lashermes, P. and Bertrand, B. (2011), Genomic expression dominance in the natural allopolyploid Coffea arabica is massively affected by growth temperature, New Phytol., 192, 760–74. doi:10.1111/j.1469-8137.2011.03833.x.
- Barre, P., Layssac, M., D’Hont, A., Louarn, J., Charrier, A., Hamon, S. and Noirot, M. (1998), Relationship between parental chromosomic contribution and nuclear DNA content in the coffee interspecific hybrid C. pseudozanguebariae × C. liberica var ‘dewevrei’, Theor. Appl. Genet., 96, 301–05. doi:10.1007/s001220050740.
- Bennett, M. D. and Leitch, I. J. (1995), ‘Nuclear DNA amounts in angiosperms’. Ann. Bot., 76, 113–76.
- Berecha, G., Aerts, R., Vandepitte, K., Van Glabeke, S., Muys, B., Roldan-Ruiz, I. and Honnay, O. (2014), ‘Effects of forest management on mating patterns, pollen flow and intergenerational transfer of genetic diversity in wild Arabica coffee (Coffea arabica L.) from Afromontane rainforests’, Biol. J. Linn. Soc. Lond., 112, 76–88. doi:10.1111/bij.12274.
- Berthaud, J. (1980), L’incompatibilite chez Coffea canephora: methode de test et determinisme genetique, Cafe Cacao The, 24, 267–74.
- Bertrand, B., Bardil, A., Baraille, H., Dussert, S. and et al. (2015), The greater phenotypic homeostasis of the allopolyploid Coffea arabica improved the transcriptional homeostasis over that of both diploid parents, Plant Cell. Physiol., 56(10), 2035–51. doi:10.1093/pcp/pcv117.
- Bouharmont, J. (1959), Recherches sur les affinites chromosomiques dans le genre Coffea, Publ. INEAC, Serie Scientifique, 77, 94.
- Bremer, B. and Eriksson, T. (2009), Time tree of Rubiaceae: phylogeny and dating the family, subfamilies, and tribes, Int. J. Plant Sci., 170, 766–93.
- Bremer B. and Jansen, R. K. (1991), Comparative restriction site mapping of chloroplast DNA implies new phylogenetic relationships within Rubiaceae, Am. J. Bot., 78, 198–213.
- Bridson, D. (1982), Studies in Coffea and Psilanthus (Rubiaceae subfam. Cinchonoideae) for Part 2 of ‘Flora of Tropical East Africa’: rubiaceae, Kew Bull., 36, 817–59.
- Bridson, D. (1987), Nomenclatural notes on Psilanthus, including Coffea sect. Paracoffea (Rubiaceae tribe Coffeeae), Kew Bull., 42, 453–60.
- Bunn, C., Laderach, P., Ovalle-Rivera, O. and Kirschke, D. (2015) ‘A bitter cup: climate change profile of global production of Arabica and Robusta coffee’, Clim. Change, 129, 89–101. doi.org/10.1007/s10584-014-1306-x.
- Cenci, A., Combes, M. C. and Lashermes, P. (2010), Comparative sequence analyses indicate that Coffea (Asterids) and Vitis (Rosids) derive from the same paleo-hexaploid ancestral genome, Molecular Genetics and Genomics, 283, 493–501. doi:10.1007/s00438-010-0534-7.
- Cenci, A., Combes, M. C. and Lashermes, P. (2012), Genome evolution in diploid and tetraploid Coffea species as revealed by comparative analysis of orthologous genome segments, Plant Mol. Biol., 78, 135–45. doi:10.1007/s11103-011-9852-3.
- Cenci, A., Combes, M. C. and Lashermes, P. (2013), Differences in evolution rates among eudicotiledon species observed by analysis of protein divergence, J. Heredity, 104(4), 459–64. doi:10.1093/jhered/est025.
- Combes, M. C., Dereeper, A., Severac, D., Bertrand, B. and Lashermes, P. (2013), Contribution of subgenomes to the transcriptome and their intertwined regulation in the allopolyploid Coffea arabica L. grown at contrasted temperatures, New Phytol., 200, 251–60. doi:10.1111/nph.12371.
- Combes, M. C., Hueber, Y., Dereeper, A., Rialle, S., Herrera, J. C. and Lashermes, P. (2015) Regulatory divergence between parental alleles determines gene expression patterns in hybrids, Genome Biol. Evol., 7(4), 1110–21. doi:10.1093/gbe/evv057.
- Coulibaly, I., Noirot, M., Lorieux, M., Charrier, A., Hamon, S. and Louarn, J. (2002), Introgression of self-compatibility from Coffea heterocalyx to the cultivated species Coffea canephora, Theor. Appl. Genet., 105, 994–9.
- Cros, J., Combes, M. C., Trouslot, P., Anthony, F., Hamon, S., Charrier, A. and Lashermes, P. (1998), Phylogenetic relationships of Coffea species: new evidence based on the chloroplast DNA variation analysis, Mol. Phylo. Evol., 9, 109–17.
- Davis, A. P., Gole, T. W., Baena, S. and Moat, J. (2012), The impact of climate change on indigenous arabica coffee (Coffea arabica): predicting future trends and identifying priorities, Plos One, 7, e47981.
- Davis, A. P., Govaerts, R., Bridson, D. M. and Stoffelen, P. (2006), An annotated taxonomic conspectus of the genus Coffea (Rubiaceae), Bot. J. Lin. Soc., 152, 465–512.
- Davis, A. P., Tosh, J., Ruch, N. and Fay, M. (2011), Growing coffee: Psilanthus (Rubiaceae) subsumed on the basis of molecular and morphological data; implications for the size, morphology, distribution and evolutionary history of Coffea, Bot. J. Lin. Soc., 167, 357–77.
- De Beenhouwer, M., Muleta, D., Peeters, B., Van Geel, M., Lievens, B. and Honnay, O. (2015),
DNA pyrosequencing evidence for large diversity differences between natural and managed coffee mycorrhizal fungal communities, Agron. Sustain. Dev., 35, 241–9. doi:10.1007/s13593-014-0231-8. - De Carvalho, K., Petkowicz, C. L. O., Nagashima, G. T., Bespalhok Filho, J. C., Vieira, L. G. E., Pereira, L. F. P. et al. (2014), Homeologous genes involved in mannitol synthesis reveal unequal contributions in response to abiotic stress in Coffea arabica, Mol. Genet. Genomics, 289, 951–63.
- Denoeud, F., Carretero-Paulet, L., Dereeper, A. and et al. (2014) The coffee genome provides insight into the convergent evolution of caffeine biosynthesis, Science, 345, 1181–4. doi:10.1126/science.1255274.
- Doyle, J. J. and Egan, A. N. (2010), Dating the origins of polyploidy events, New Phytol., 186, 73–85.
- Gomez, C., Dussert, S., Hamon, P., Hamon, S., Kochko, A. and Poncet, V. (2009), Current genetic differentiation of Coffea canephora Pierre ex A. Froehn in the Guineo-Congolian African zone: cumulative impact of ancient climatic changes and recent human activities, BMC Evol. Biol., 9, 167. doi:10.1186/1471-2148-9-167.
- Goncalves, M. M. and Rodrigues, M. L. (1976), Estudos sobre o cafe de Timor. II. Nota sobre as posibilidades de producao do hibrido de Timor no seu habitat natural, Lisboa, Portugal. Missao de Estudos Agronomicos do Ultramar, Portugal. Comunicacoe, 86, 31–72.
- Guyot, R., Darre, T., Dupeyron, M., de Kochko, A., Hamon, S., Couturon, E., Crouzillat, D., Rigoreau, M., Rakotomalala, J. J., Raharimalala, N. E., Akaffou, S. D. and Hamon, P. (2016), Partial sequencing reveals the transposable element composition of Coffea genomes and provides evidence for distinct evolutionary stories, Mol. Genet. Genomics, 291(5), 1979–90.
- Hamon P., Grover CE., Davis AP., Rakotomalala JJ., Raharimalala NE. et al. (2017), Genotyping-bysequencing provides the first well-resolved phylogeny for coffee (Coffea) and insights into the evolution of caffeine content in its species, Mol. Phylo. Evol., 109, 351–361.
- Jackson, S. and Chen, Z. J. (2010), Genomic and expression plasticity of polyploidy, Curr. Opin. Plant Biol., 13, 153–9.
- Kiehn, M. (1995), Chromosome survey of the Rubiaceae, Ann. Missouri Bot. Gard., 82, 398–408.
- Krug C. A. and Mendes A. J. T. (1940), Cytological observations in Coffea - IV, J. Genet., 39, 189–203.
- Lashermes, P., Combes, M. C., Hueber, Y., Severac, D. and Dereeper, A. (2014), Genome rearrangements derived from homoeologous recombination following allopolyploidy speciation in coffee, Plant J., 78, 674–685. doi:10.1111/tpj.12505.
- Lashermes, P., Combes, M. C., Robert, J., Trouslot, P., D’Hont, A., Anthony, F. and Charrier, A. (1999),
Molecular characterisation and origin of the Coffea arabica L. genome, Mol. Gen. Genet., 261, 259–66. - Lashermes, P., Combes, M. C., Trouslot, P. and Charrier, A. (1997), Phylogenetic relationships of coffee tree species (Coffea L.) as inferred from ITS sequences of nuclear ribosomal DNA, Theor. Appl. Genet., 94, 947–55.
- Lashermes, P., Couturon, E., Moreau, N., Paillard, M. and Louarn, J. (1996), Inheritance and genetic mapping of self-incompatibility in Coffea canephora Pierre, Theor. Appl. Genet., 93, 458–62.
- Lashermes, P., Cros, J., Combes, M. C., Trouslot, P., Anthony, F., Hamon, S. and Charrier, A. (1996), Inheritance and restriction fragment length polymorphism of chloroplast DNA in the genus Coffea L., Theor. Appl. Genet., 93, 626–32.
- Lashermes, P., Hueber, Y., Combes, M. C., Severac, D. and Dereeper, A. (2016), Inter-genomic DNA exchanges and homoeologous gene silencing shaped the nascent allopolyploid coffee genome (Coffea arabica L.), G3 Genes Genom. Genet., 6, 2937–48. doi:10.1534/g3.116.030858.
- Lashermes, P., Paczek, V., Trouslot, P., Combes, M. C., Couturon, E. and Charrier, A. (2000), Singlelocus inheritance in the allotetraploid Coffea arabica L. and interspecific hybrid C. arabica x C. canephora, J. Heredity, 91, 81–5.
- Madlung, A. and Wendel, J. F. (2013), Genetic and epigenetic aspects of polyploid evolution in plants, Cytogenet. Genome Res., 140, 270–85.
- Mahe, L., Le Pierres, D., Combes M. C. and Lashermes, P. (2007), Introgressive hybridization between the allotetraploid Coffea arabica and one of its diploid ancestors, Coffea canephora, in an exceptional sympatric zone in New Caledonia, Genome, 50, 316–24.
- Marraccini, P., Freire, L. P., Alves, G. S. C., Vieira, N. G., Vinecky, F., Elbelt, S. and et al. (2011), RBCS1 expression in coffee: Coffea orthologs, Coffea arabica homeologs, and expression variability between genotypes and under drought stress, BMC Plant Biol., 11, 85.
- Maurin, O., Davis, A. P., Chester, M., Mvungi, E. F., Jaufeerally-Fakim, Y. and Fay, M. F. (2007), Towards a phylogeny for Coffea (Rubiaceae): identifying well-supported lineages based on nuclear and plastid DNA sequences, Ann. Bot., 100, 1565–83.
- Mehrabi, Z. and Lashermes, P. (2017), Protecting the origins of coffee to safeguard its future, Nature Plants, 3, 16209. doi:10.1038/nplants.2016.209.
- Noirot, M., Charrier, A., Stoffelen, P. and Anthony, F. (2016), Reproductive isolation, gene flow and speciation in the former Coffea subgenus: a review, Trees, 30, 597. doi:10.1007/s00468-015-1335-8.
- Noirot, M., Poncet, V., Barre, P., Hamon, P., Hamon, S. and de Kochko, A. (2003) Genome size variations in diploid African Coffea species, Ann. Bot., 92(5), 709–14.
- Nowak, M. D., Davis, A. P. and Yoder, A. D. (2012), Sequence data from new plastid and nuclear COSII regions resolves early diverging lineages in Coffea (Rubiaceae), Syst. Bot., 37, 995–1005.
- Nowak, M. D., Davis, A. P., Anthony, F. and Yoder, A. D. (2011), Expression and Trans-Specific Polymorphism of Self-Incompatibility RNases in Coffea (Rubiaceae), PLoS One, 6(6), e21019. doi:10.1371/journal.pone.0021019.
- Pinto-Maglio, C. A. F. and Da Cruz, N. D. (1998), Pachytene chromosome morphology in Coffea L. II. C. arabica L. complement, Caryologia, 51, 19–35.
- Pinto-Maglio, C. A. F. (2006), Cytogenetics of coffee, Braz. J. Plant Physiol., 18(1), 37–44.
- Raina S. N., Mukai, Y. and Yamamoto, M. (1998), In situ hybridisation identifies the diploid progenotor of Coffea arabica (Rubiaceae), Theor. Appl. Genet., 97, 1204–9.
- Razafinarivo, N. J., Rakotomalala, J. J., Brown, S. C., Bourge, M., Hamon, S., de Kochko, A., Poncet, V., Dubreuil-Tranchant, C., Couturon, E., Guyot, R. and Hamon, P. (2012), Geographical gradients in the genome size variation of wild coffee trees (Coffea) native to Africa and Indian Ocean islands, Tree Genet. Genomes, 8, 1345–58.
- Robbrecht, E. and Manen, J. F. (2006), The major evolutionary lineages of the coffee family (Rubiaceae, angiosperms). Combined analysis (nDNA and cpDNA) to infer the position of Coptosapelta and Luculia, and supertree construction based on rbcL, rps16, tnrL-trnF, and atpB-rbcL data. A new classification in two subfamilies, Cinchonoideae and Rubioideae, Syst. Geogr. Pl., 76, 85–146.
- Soltis, D. E., Soltis P. S. and Tate, J. A. (2003), Advances in the study of polyploidy since plant speciation, New Phytol., 161, 173–91.
- Song, Q. and Chen, Z. J. (2015), Epigenetic and developmental regulation in plant polyploids, Curr. Opin. Plant. Biol., 24, 101–9
- Stoffelen, P., Noirot, M., Couturon, E., Bontems, S., De Block, P. and Anthony, F. (2009), Coffea anthonyi, a new self-compatible Central African coffee species, closely related to an ancestor of Coffea arabica, Taxon, 58, 133–40.
- Tesfaye, K., Govers, K., Bekele, E. and Borsch, T. (2014), ISSR fingerprinting of Coffea arabica throughout Ethiopia reveals high variability in wild populations and distinguishes them from landraces, Plant Syst. Evol., 300, 881–97. doi:10.1007/s00606-013-0927-2.
- Thomas, A. S. (1942), The wild arabica coffee on the Boma Plateau, Anglo-Egyptian Sudan, Emp. J. Exp. Agric., 10, 207–12.
- Thomas, A. S. (1944), The wild coffees of Uganda, Emp. J. Exp. Agric., 45, 1–12.
- Van der Vossen, H. A. M., Bertrand, B. and Charrier, A. (2015) Next generation variety development for sustainable production of arabica coffee (Coffea arabica L.): a review, Euphytica, 204(2), 243–25. doi:10.1007/s10681-015-1398-z.
- Woldermariam, G. T., Denich, M., Teketay, D. and Vlek, P. L. G. (2002),
Human impacts on the Coffea arabica genepool in Ethiopia and the need for its in situ conservation.
In Brown, A. H. D., Jackson, M. T., Engels, J. M. M., Ramantha Rao, V. (Eds), Managing Plant Genetic Diversity. IPGRI: Rome, Italy, pp. 237–47.
'Coffee Genetics' 카테고리의 다른 글
| 세포핵 내 유전체 접힘의 3차원 구조 (0) | 2024.07.31 |
|---|---|
| 아라비카 커피의 염색체-규모 어셈블리 - 染色體 異常 및 交換 (0) | 2024.07.24 |
| Oxford Nanopore Technologies Sequencing - 3세대 시퀀싱 (1) | 2024.07.21 |
| Complete Genomics cPAL Sequencing - 3세대 시퀀싱 (0) | 2024.07.19 |
| PacBio SMRT Sequencing - 3세대 DNA 시퀀싱 (0) | 2024.07.17 |




댓글